当前位置:网站首页>单细胞论文记录(part14)--CoSTA: unsupervised convolutional neural network learning for ST analysis
单细胞论文记录(part14)--CoSTA: unsupervised convolutional neural network learning for ST analysis
2022-06-22 05:45:00 【GoatGui】
学习笔记,仅供参考,有错必纠
Authors:Yang Xu, Rachel Patton McCord
Journal:BMC Bioinformatics
Year:2021
Keywords: Spatial transcriptomics, Gene clustering, Convolutional neural network
文章目录
CoSTA: unsupervised convolutional neural network learning for spatial transcriptomics analysis
Abstract
Background: 空间转录组学技术的兴起,使人们对基因调控(gene regulation)如何在空间背景下发生有了新的认识. 确定哪些基因以类似的空间模式表达,可以揭示一个组织中不同类型细胞的基因调控关系. 然而,目前许多分析方法没有充分利用数据的空间组织,而是将pixels作为独立的特征. 在此,我们提出了CoSTA:一种通过卷积神经网络(ConvNet)聚类学习基因表达矩阵之间空间相似性的新方法.
Results: 通过分析模拟的和以前发表的空间转录组学数据,我们证明CoSTA以强调更广泛的空间模式而不是pixel-level相关的方式学习基因之间的空间关系. CoSTA提供了每对基因之间表达模式相似性的定量测量,而不仅仅是将基因归类. We find that CoSTA identifies narrower, but biologically relevant, sets of significantly related genes as compared to other approaches.
Conclusions: 深度学习的CoSTA方法通过关注表达模式的shape 为空间转录组学分析提供了一个不同的角度,与overlap or pixel correlation approach,它使用了更多关于相邻pixels的位置信息. CoSTA可以应用于任何以矩阵形式表示的空间转录组学数据,并可能在未来应用于组织学等数据集,其中不同基因的图像来自相似但不相同的生物切片.
Background
空间转录组学最近得到了科学界的广泛关注. 不同的技术已经能够高分辨率地测量基因调控是如何在一个组织或数千个单细胞的空间组织中进行[1]. 对这些数据的分析有可能揭示出基因之间的空间调控关系. 然而,目前的分析pipelines 往往将表达矩阵中的每个像素作为一个独立的特征,从而失去了空间信息. 例如,seqFISH+技术可以在单细胞分辨率下原位荧光检测10,000个mRNA,而且经常有一些细胞群与它们的邻居的基因表达相关联,组成更大的结构. 然而,以往的文章使用PCA和分层聚类来分析这些表达模式,将每个细胞作为一个独立的特征,而不是保留细胞邻居的空间位置[2]. Slide-seq同样产生高通量的空间分辨率转录信息,使用sequencing 而不是fluorescence. 以前对Slide-seq数据的分析首先确定了空间上的非随机基因表达,但随后使用像素级的重叠分析而不是根据空间特征来寻找表达相似模式的基因[3]. 现有的空间转录组学分析算法是基于统计建模的,主要提出将空间表达或可变(SE或SV)的基因与随机的空间表达噪音区分开来. 例如,SpatialDE和SPARK分析方法都估计了基因的空间模式的重要性[4, 5]. SpatialDE进一步建立了一个无监督检测算法,将有意义的SE基因聚类到不同的群体中,这些群体具有一定的空间模式. 相比之下,SPARK只为寻找SE基因而设计. 为了检查基因之间的空间关系,该方法仍然依赖于使用单个像素作为特征的层次化聚类. 因此,即使SPARK能识别具有显著空间模式的基因,SPARK分析的后半部分也会将表达与它的原始空间背景脱钩. 到目前为止,现有的空间转录组学分析要么涉及多步骤的复杂特征工程的空间量化,要么涉及人为的硬性规定或基于统计建模的候选SE基因的筛选. 在现有的方法中,两个基因之间的表达模式的相似性要么是二元的(基因是否聚集在一起),要么是基于pixel-level的相关度进行量化.
在这项工作中,我们提出了一种受计算机视觉和图像分类启发的方法,以寻找不同基因的空间表达模式之间的关系,同时保留完整的空间背景(图1a). 我们的目标是以保留相邻细胞和组织区域之间空间关系的方式找到基因表达模式之间的定量比较. 即使某些像素组不完全重叠,也能识别出整体类似的表达形状. 这在概念上类似于计算机视觉任务中的图像识别. 卷积神经网络的使用带来了计算机视觉中深度学习的成功,并展示了广泛的应用,包括图像分类和物体识别. 一些小组提出了不同的方法来使用卷积神经网络(ConvNet)进行无监督学习[6-8]. 因此,在这里,我们采用了一种无监督的ConvNet学习策略,用于空间转录组学分析(CoSTA). 通过模拟数据,我们表明CoSTA可以正确分类各种不同的空间模式,而且CoSTA所检测的模式取决于空间分组而不是单个像素. 然后,我们将CoSTA应用于已发表的MERFISH和Slide-seq数据,并表明CoSTA有时会识别出空间关系的较小的基因组,但这些被识别的关系是生物学上的.

Results
CoSTA architecture: training a ConvNet with pseudo‑labels generated by GMM clustering
虽然有很多无监督学习策略,但我们选择应用DeepCluster的工作流程,因为它是直接和容易实现的[6]. 我们的CoSTA方法包括两个主要部分:通过高斯混合模型(GMM)进行聚类,以及训练神经网络时通常进行的权重更新(图1a,详细说明见方法).
我们的输入是一组基因表达图像,其中每个图像是记录一个基因在空间每个位置的表达水平的矩阵,所有图像都属于同一个生物空间. 我们首先随机地初始化一个ConvNet,然后通过ConvNet转发这些基因表达矩阵. 我们的ConvNet由三个卷积层组成,每个卷积层后面都有一个批量归一化层和一个最大池化层. 我们将最后一个最大池化层的矩阵输出平铺成一个向量,以捕捉基因表达数据的空间特征. 这个向量的大小将根据特定空间转录组学技术的图像大小而变化. 然后,在对基因进行GMM聚类之前,我们在各特征间应用L2归一化,并通过UMAP降低维度. 在降维过程中,UMAP可以保留全局和局部结构,并且以前在图像聚类中显示出比其他降维方法如Isomap和t-SNE更好的性能[7, 9]. 这种聚类的目的是为了产生标签,以便我们能够像其他常见的监督神经网络训练方法那样更新ConvNet. 当ConvNet被随机初始化时,这个ConvNet所提取的特征是很弱的. 然而,使用它们生成标签仍然可以引导ConvNet学习更多的特征. 事实上,Caron等人表明DeepCluster可以从弱信号中学习,以引导ConvNet的判别能力[6]. 我们没有给每个基因一个单一的聚类标签,而是将一个辅助的目标分布作为一个软分配. 这种方法强调了在聚类任务中具有高置信度的基因,并且不考虑从ConvNet的随机初始化中持续存在的嘈杂标签. 这样做也可以为训练神经网络带来更稳定的目标值[8]. 最后,我们使用这些软分配来训练ConvNet. 我们在ConvNet之后添加一个全连接层,为每个基因分配到每个标签产生概率. 因此,我们可以通过最小化基于GMM聚类的软分配和全连接层的概率之间的bi-tempered logistic loss based on Bregman Divergences来优化模型[10].
总之,CoSTA方法使用ConvNet聚类结构,重复以下3个过程:
- 通过ConvNet生成特征
- 通过GMM聚类生成软分配
- 使用软分配来更新ConvNet
一旦我们完成了训练,我们只保留训练好的ConvNet,用于特征提取的目的. 由于ConvNet主要由卷积层组成,ConvNet提取的每个基因的最终向量应该是一个空间表示. 使用这个空间表示,我们就可以在一个空间转录组学数据集中量化任何两个基因之间的关系,通过UMAP将这个数据集中的所有SE基因可视化,并通过常见的聚类算法分配patterns. 关于这个学习架构的原理的更多细节可以在文章的Methods部分找到.
Rationale for using spatial patterns rather than exact pixel overlap
为了证明重叠分析所损失的空间信息,以及为什么像CoSTA这样的空间表示方法是有用的,我们提出了一个简化的生物启发的概念性例子(图1b).
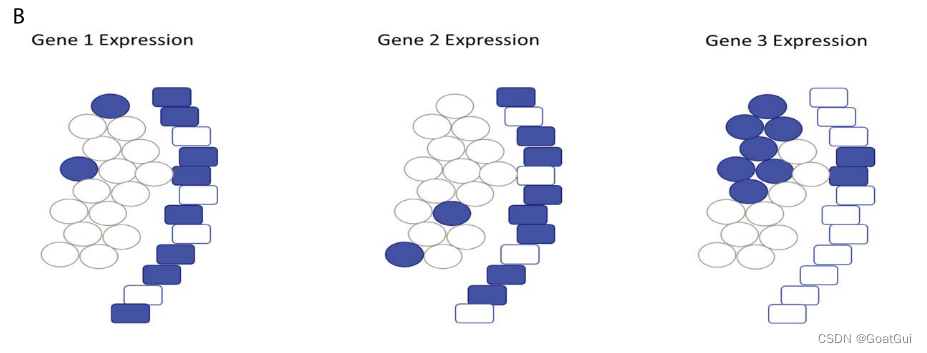
在生物组织切片中,我们通常观察到一些结构,如紧密连接的上皮细胞层(图中的矩形)与基质细胞(圆圈)的集合相邻. 在这个例子中,显示了三个基因的空间表达模式. 通过重叠比较基因表达模式,我们观察到基因1和2的重叠量与基因1和3的重叠量相同(40%). 因此,用重叠的方法来衡量基因模式的相似性,就像以前Slide-seq分析中使用的方法,会报告说基因1与基因2和基因3同样相似[3]. 然而,在生物学上,基因1和基因2主要在上皮层中表达,而基因3在基质中表达. 这种生物学上的差异不是通过严格的重叠来检测的,而是需要一种空间表示,将检测上皮层表达的垂直条纹作为一种突出的模式. 在计算机视觉中,过滤器通常被用来寻找这种局部关联,ConvNet在模式识别中的成功也依赖于使用过滤器来识别局部关联. 利用这3个基因对相同过滤器的反应信号,ConvNet方法将识别出基因1和2更相似,基因3更不相似. 因此,我们有动力使用我们基于ConvNet聚类的CoSTA方法来优先考虑相似的形状,而不是重叠的生物案例,在这些案例中,细胞层和细胞组的整体模式比独立的单个细胞更重要[11].
Tests on synthetic data show CoSTA’s high specificity, reliance on spatial relationships, and ability to distinguish signal from noise
作为对CoSTA在没有完全重叠的情况下检测correlated spatial patterns能力的第一次测试,我们使用了MNIST手写数字图像数据[12]. 当目的是找到哪些数字与数字3有相关的手写模式时,CoSTA只识别出数字3的其他实例是相关的(100% specificity). 相反,重叠分析发现所有其他数字的一些样本是3的相关数字(58% specificity). 同时,CoSTA将数字3 s的更小的子集识别为相关数字(35% sensitivity),而重叠分析在其较少的特异性集合中捕捉到更多的相关数字(65% sensitivity). 如下图所示,this increased specificity but possibly decreased sensitivity of CoSTA compared to other techniques appears to hold true in biological data as well.
在将CoSTA应用于真实的空间转录组学数据之前,我们接下来在5个合成数据集上测试其性能,这些数据集是根据小鼠嗅球的真实表达模式模拟的,遵循SPARK的模拟方法(图1c左栏) [5, 13]. 我们为每个模式生成了2000个假的基因表达矩阵,以模拟10,000个总基因的数据. 为了模拟每个基因的噪声和变异性,我们根据均值为0、方差为0.2至0.6的正态分布,在每个空间坐标上独立添加了残余误差. 然后,我们评估了CoSTA是否能将每个模拟的噪声基因分配到正确的模式. 为了将CoSTA得出的聚类分配与真实标签进行比较,我们使用了聚类评价指标归一化互信息(NMI)[14]. 当基因分配到5个模式中变得越来越准确时,NMI的值就接近1. 当CoSTA初始化时,NMI在0.27到0.57之间(图1c右栏). 随着训练的进行,CoSTA学会了区别5种模式的特征,最终实现了对真实类别标签的NMI从0.85到0.98(图1c右面板). 对于最高的噪声水平(0.6),我们发现在CoSTA训练期间,结合center loss (CL)和bi-tempered logistic loss,大大改善了CoSTA的准确性(NMI从0.52增加到0.91). 然而,center loss将样本推向5个中心点,并且只适用于已知最终模式数量的情况. 因此,我们不包括真实生物情况下的CL.

为了证明CoSTA从这些合成数据集中学习了空间模式而不是像素级模式,我们对这些合成数据集中的像素位置进行shuffling. 以完全相同的方式shuffle所有的基因矩阵,使像素上的重叠信息保持一致,同时破坏了相邻像素之间的相关性,从而破坏了空间模式. 如果一个模式检测方法成功地利用了相邻像素之间的空间关系,那么它对模式的分类能力就应该被这种shuffling所破坏. 事实上,我们发现CoSTA不能像shuffling数据那样将基因区分为正确的模式标签(NMI从0.32到0.89不等),表明CoSTA检测的是取决于相邻像素位置的空间特征,而不是可以由一组单一像素捕捉的特征. 当我们将在0.4噪音水平下训练的CoSTA模型应用于逐渐增多的shuffling图像时,我们发现将基因分类为组的能力与shuffling量成比例下降. 我们还在这些真实和shuffling的合成数据集上测试了SpatialDE. SpatialDE在真实数据集上的表现非常好,正如预期. 然而,shuffling数据通常不会改变SpatialDE的性能,表明CoSTA和SpatialDE之间的一个重要区别:SpatialDE更可能检测单个像素的模式,而CoSTA强调这些像素相对于彼此的空间位置和模式的整体形状.
使用这个相同的合成数据,我们接下来进行了一个干扰测试,以证明使用单个像素作为特征来分析空间转录组学数据的一个缺点. 对于一半的模拟基因矩阵,我们掩盖了图案的某个区域,被掩盖的区域在视觉上不会改变表达模式. 这模拟了在实验中由于技术原因某个区域被遮挡或不能很好地采样的情况. 在这种情况下,使用像素重叠来识别模式,将被掩盖和未被掩盖的基因分配到不同的组中,即使它们属于同一模式. 相比之下,CoSTA对这种干扰有抵抗力.
在真正的空间转录组学数据中,并不是所有的基因都会属于一个明确的空间模式–一些与给定组织或条件无关的基因可能只产生随机噪音或相当均匀地表达. 为了模仿这种情况,我们进一步遵循SPARK中的模拟方法,生成具有5种空间模式的合成数据集,并有混合的SE(空间表达)和非SE基因. 我们在这些数据上训练CoSTA,SE和非SE基因的比例不同,从90:10到10:90. 我们发现CoSTA对SE基因的表述与非SE基因截然不同,即使CoSTA是用高比例的非SE基因进行训练. 同时,即使存在非SE基因,CoSTA也表现出区分SE基因的不同模式的能力. 此外,CoSTA甚至没有将大量的非SE基因分成独立的类别,表明它不会从噪声中产生错误的信号. 在此,我们还注意到,与SpatialDE等方法相比,CoSTA的一个优势是输出的特征向量能够实现可视化,正如这些模拟结果中所呈现的那样. 虽然Spatial DE可以将基因分类,但它产生的结果并不能像我们这里的CoSTA那样,能够直观地看到SE和非SE基因是如何分开的. 总的来说,CoSTA在合成数据方面的表现表明,CoSTA可以学习辨别的空间特征.
边栏推荐
- U disk as startup disk to reinstall win10 system (no other software required)
- 自控原理之系统辨识
- 401-字符串(344. 反转字符串、541. 反转字符串II、题目:剑指Offer 05.替换空格、151. 颠倒字符串中的单词)
- Market consumption survey and investment prospect forecast report of China's graphite industry 2022-2027
- Frame profiling
- Machine learning Note 6: number recognition of multiple classification problems in logistic regression
- idea插件Easy Code的简单使用
- n个整数的无序数组,找到每个元素后面比它大的第一个数,要求时间复杂度为O(N)
- 单球机器人动力学与控制研究
- Server PHP related web page development environment construction
猜你喜欢

活动预告|EdgeX 开发者峰会@南京站 来啦!

Machine learning Note 6: number recognition of multiple classification problems in logistic regression

Creating GLSL Shaders at Runtime in Unity3D

Ethernet UDP frame contract design

Frame profiling
AUTOSAR from getting started to mastering 100 lectures (150) -soa architecture and Application

Single precision, double precision and precision (Reprint)

idea插件Easy Code的简单使用

MFC TabCtrl 控件修改标签尺寸

Understanding of C pointer
随机推荐
Simple use of idea plug-in easy code
EPP (enhanced parallel port)
AUTOSAR from getting started to mastering 100 lectures (150) -soa architecture and Application
Ethernet communication protocol
JTAG interface
Unity encrypts ASE game data
组合逻辑块的测试平台
Improve your game‘s performance
单精度,双精度和精度(转载)
Reptile initial and project
printf becomes puts
I2C interface
Record some problems and solutions encountered in processing SIF data
Market development trend forecast and investment risk outlook report of China's silicon carbide industry 2022-2027
Case analysis of terminal data leakage prevention
性能优化最佳实践之缩减游戏大小
电热水壶坏了别扔,它很容易修好的!
DOS bat syntax record I
富设备平台突破:基于RK3568的DAYU200进入OpenHarmony 3.1 Release主干
Machine learning note 7: powerful neural network representation