当前位置:网站首页>使用shapeit进行单倍型分析
使用shapeit进行单倍型分析
2022-06-21 09:36:00 【51CTO】
欢迎关注”生信修炼手册”!
shapeit是一款单倍型分析工具,运算速度快,准确率高,是impute2官方推荐的pre-phasing工具,官网如下
http://mathgen.stats.ox.ac.uk/genetics_software/shapeit/shapeit.html
对应的文献发表在nature method上,链接如下
通过隐马可夫模型来分析单倍型,简化的模型示意如下

从上到下依次有5个子图,用1到5来表示,需要分成3个部分来看。在1图中,表示的是8个位点构成的8种单倍型,每行表示一个单倍型,每一列代表一个位点,2图中用图状结构来表示上述的单倍型,每个节点表示一个SNP位点,依次用Z1到Z8表示,从1到8的完整路径代表一个单倍型。观察图1可以发现,前4个位点只有3种组成,后4个位点也是同样的,通过4号位点和5号位点的不同连线可以表示所有的单倍型,每条边上的数字代表对应的频数。
图5表示的是某个样本的分型结果,分别用0,1,2表示不同的状态,0表示没有突变,1表示杂合突变,2表示纯合突变,根据分型结果拆分成单倍型的时候,杂合突变对应2个allel, 根据这个分型结果可以得到图4中对应的单倍型构成,图4中ref allel用空白方框表示,alt allel用黑色方框表示,对于前5个位点,存在了2个杂合突变,所以有4种路径,后3个位点也是4种。
图3表示的是该软件的隐马可夫模型,将真实的单倍型当做是隐藏序列,将根据分型结果预测的单倍型当做是观察序列,通过建模之后来分析隐藏序列的组成,就得到了真实的单倍型分析结果。
在文献中,将该软件与其他类似的工具进行了比较,结果示意如下
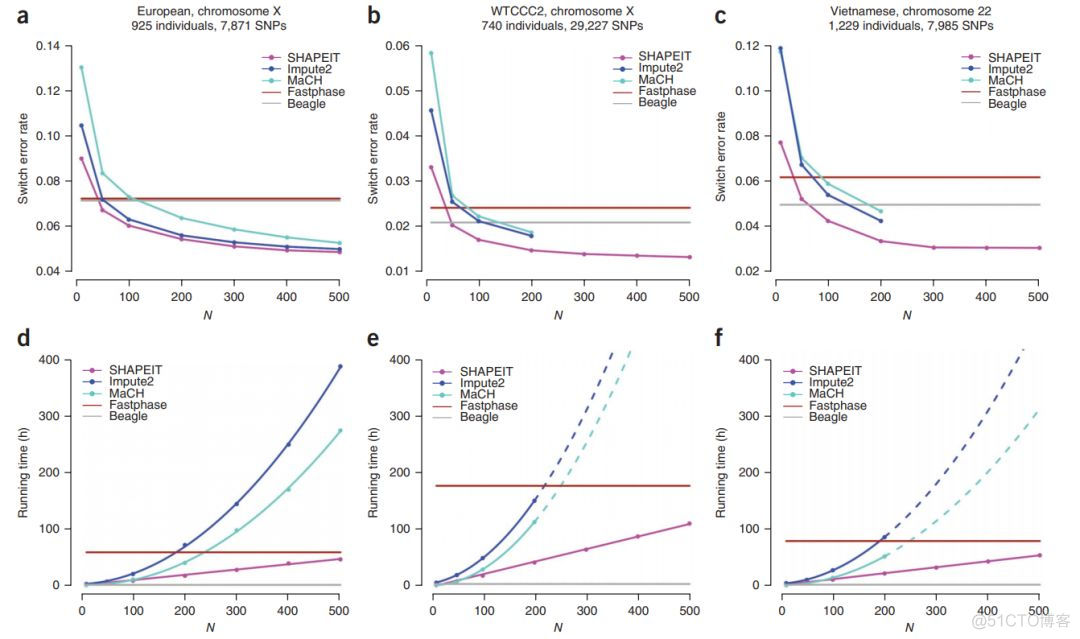
采用了3个不同的数据集,比较了运行时间和错误率,shapeit错误率最低,运行速度最快。
该软件的基本用法如下
需要指定的参数分成了以下3个部分
1. input unphased genotypes
支持以下3种格式
- ped/map
- bed/bim/fam
- gen/sample
- vcf
前两种为plink软件的格式,是GWAS分析最常见的文件格式,第三种格式是WTCCC默认的文件格式 第四种是最常见的VCF格式。
不同类型的输入文件对应的用法如下
shapeit \
--input-ped gwas.ped gwas.map \
-M genetic_map.txt \
--missing-code N \
-O gwas.phased
shapeit \
--input-bed gwas.bed gwas.bim gwas.fam \
-M genetic_map.txt \
-O gwas.phased
shapeit \
--input-gen gwas \
-M genetic_map.txt \
-O gwas.phased
shapeit \
--input-vcf gwas.vcf \
-M genetic_map.txt \
-O gwas.phased
- 1.
- 2.
- 3.
- 4.
- 5.
- 6.
- 7.
- 8.
- 9.
- 10.
- 11.
- 12.
- 13.
- 14.
- 15.
- 16.
- 17.
- 18.
- 19.
- 20.
对于gen/sample文件格式,可以通过gtool这个软件来进行格式转换,
2. genetic map
参考基因组对应的连锁图谱,可以提高单倍型分析的准确性,官方提供了hapmap项目的连锁图谱供下载,链接如下
http://mathgen.stats.ox.ac.uk/genetics_software/shapeit/shapeit.html#formats
这个是一个可选参数,没有的情况下软件会根据线性模型来进行估算。
3. output phased haplotypes
默认用后缀为haps和sample的两个文件来描述单倍型, haps文件的内容如下所示

每列之间用空格分隔,第一列为snp位点所在的染色体名称,第二列为snp id,第三列为染色体的位置,第四列为不同样本中该位点的分型结果,0代表ref allle, 1代表alt allel, 每两列对应一个样本。
后缀为sample的文件内容如下所示
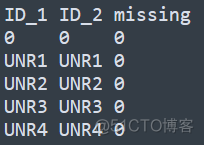
用来描述样本的信息,同样的空格分隔,前两行内容固定,后续每一行代表一个样本,以上只是该文件最基本内容的展示,还可以有更多的列,用来描述样本的表型信息。
在impute2中,phased reference panel会用hap/legend/sample3个文件来表示,通过下列代表可以进行格式转换
不同格式的详细解释参考以下链接
http://mathgen.stats.ox.ac.uk/genetics_software/shapeit/shapeit.html#formats
事先对需要填充的样本进行phasing, 可以有效提高填充的运行效率,如果后续使用impute2进行基因型填充,推荐使用shapeit对需要填充的样本进行单倍型分析。
·end·
—如果喜欢,快分享给你的朋友们吧—
扫描关注微信号,更多精彩内容等着你!

边栏推荐
- The spingboot microservice is packaged into a docker image and connected to the database
- Concurrency - condition variable
- stm32mp1 Cortex M4开发篇11:扩展板蜂鸣器控制
- poi实现操作生成word表格和操作word中的图表数据
- Telecommuting Market Research Report
- Three key directories in R language and their corresponding priorities: R_ Home directory, user directory, current working directory, files read by R's startup process
- Zhihu wanzan: what kind of programmers are still wanted by the company after the age of 35? Breaking the "middle age crisis" of programmers
- Request and response must know
- Common basic functions of R language: call the data editor with edit function to manually customize and edit the data object without changing the content of the original data object, and call the data
- 嵌入式软件项目流程、项目启动说明书(示例)
猜你喜欢
![[actual combat] STM32 FreeRTOS porting series Tutorial 4: FreeRTOS software timer](/img/16/ad38288689f629106a19a0b8defea2.jpg)
[actual combat] STM32 FreeRTOS porting series Tutorial 4: FreeRTOS software timer
Stm32mp1 cortex M4 development part 10: expansion board nixie tube control
Alibaba P6 employees came to a small company for an interview and asked for an annual salary increase of 500000 yuan. How dare you speak
Lei niukesi --- basis of embedded AI

1. is god horse a meta universe?

111. solve the problem of prohibiting scripts from running on vs code. For more information, see error reporting
![[actual combat] STM32 FreeRTOS migration series tutorial 7: FreeRTOS event flag group](/img/1c/10add042271c11cd129ddfce66f719.jpg)
[actual combat] STM32 FreeRTOS migration series tutorial 7: FreeRTOS event flag group

\Processing method of ufeff

Lodash real on demand approach

TC软件概要设计文档(手机群控)
随机推荐
115. secondary packaging of table components
Introduction to ground plane in unity
The spingboot microservice is packaged into a docker image and connected to the database
Stm32mp1 cortex M4 Development Chapter 11: expansion board buzzer control
Audio and video format introduction, encoding and decoding, audio and video synchronization
How do I listen for changes in DOM element size?
2022年中总结-一步一个脚印,踩出柳暗花明
Unity VUFORIA 推荐设备
111. solve the problem of prohibiting scripts from running on vs code. For more information, see error reporting
Mobile applications introduce static Cordova according to different platforms
The way of filling holes in APK Decompilation
Float floating layout clear floating
并发编程高级部分:并行流,Tasks和Executors以及CompletableFuture类
并发-条件变量
Arcore supported devices
Wechat applet
The R language uses the sink function to export the string to the txt file in the specified directory. If no directory is specified, it will be output to the current working dir
Source insight shortcut key cross reference
121. Redux detailed summary + effect drawing + Case
Introduction to list operation in C #