当前位置:网站首页>pytorch提取骨架(可微)
pytorch提取骨架(可微)
2022-07-06 09:28:00 【深山里的小白羊】
前言
提取骨架有许多现成的包,最简单直接的就是:
from skimage.morphology import skeletonize, skeletonize_3d
但今天要介绍另外一种提取骨架的方法!也可以理解为细化
使用pytorch实现的目的是,这个过程是可微的,换言之,就可以梯度反传的,对于网络预测的mask,可以通过这个函数提取骨架,然后在骨架上约束物体的拓扑结构
该方法来自于文献CVPR 2021:clDice - a Novel Topology-Preserving Loss Function for Tubular Structure Segmentation
源码:https://github.com/jocpae/clDice
(源码非常非常的简单,这是我看到的为数不多的如此简洁的CVPR源码了)
源码
import torch
import torch.nn as nn
import torch.nn.functional as F
def soft_erode(img):
if len(img.shape)==4:
p1 = -F.max_pool2d(-img, (3,1), (1,1), (1,0))
p2 = -F.max_pool2d(-img, (1,3), (1,1), (0,1))
return torch.min(p1,p2)
elif len(img.shape)==5:
p1 = -F.max_pool3d(-img,(3,1,1),(1,1,1),(1,0,0))
p2 = -F.max_pool3d(-img,(1,3,1),(1,1,1),(0,1,0))
p3 = -F.max_pool3d(-img,(1,1,3),(1,1,1),(0,0,1))
return torch.min(torch.min(p1, p2), p3)
def soft_dilate(img):
if len(img.shape)==4:
return F.max_pool2d(img, (3,3), (1,1), (1,1))
elif len(img.shape)==5:
return F.max_pool3d(img,(3,3,3),(1,1,1),(1,1,1))
def soft_open(img):
return soft_dilate(soft_erode(img))
def soft_skel(img, iter_):
img1 = soft_open(img)
skel = F.relu(img-img1)
for j in range(iter_):
img = soft_erode(img)
img1 = soft_open(img)
delta = F.relu(img-img1)
skel = skel + F.relu(delta-skel*delta)
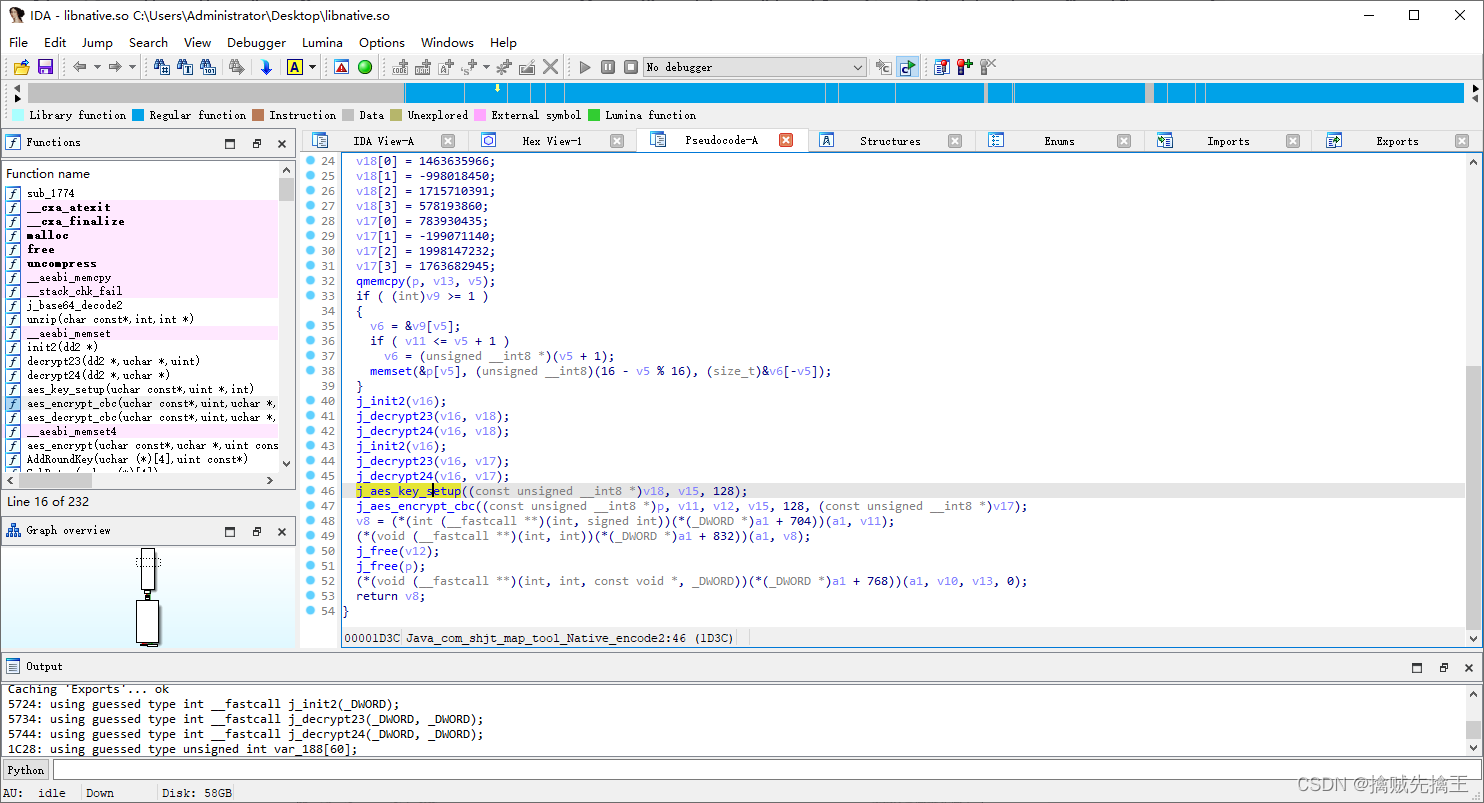
return skel
这就是文中提取骨架的所有代码了,真的非常的简洁
测试
以一张CVPPP的label为例:
为了测试提取骨架的代码,我这里以上面图像的边界为前景,来细化边界。
用下面这个函数来提取边界(同时膨胀三次):
import numpy as np
from PIL import Image
from scipy import ndimage
from skimage.segmentation import find_boundaries
def extract_boundary(label, dilation=False):
boundary = np.zeros_like(label, dtype=np.uint8)
ids, counts = np.unique(label, return_counts=True)
for i, id in enumerate(ids):
if id == 0:
boundary[label == 0] = 0
else:
tmp = np.zeros_like(label)
tmp[label == id] = 1
tmp_bound = find_boundaries(tmp != 0, mode='outer')
if dilation:
tmp_bound = ndimage.binary_dilation(tmp_bound, iterations=3, border_value=1)
boundary[tmp_bound == 1] = 1
return boundary
准备就绪,上主函数:
if __name__ == "__main__":
label = np.asarray(Image.open('plant154_label.png'))
boundary = extract_boundary(label, dilation=True)
boundary_uint8 = boundary.astype(np.uint8) * 255
Image.fromarray(boundary_uint8).save('boundary.png')
boundary = boundary.astype(np.float32)
boundary = boundary[np.newaxis, np.newaxis, ...]
boundary = torch.from_numpy(boundary)
skel = soft_skel(boundary, 10)
skel = np.squeeze(skel.numpy())
skel = (skel * 255).astype(np.uint8)
Image.fromarray(skel).save('skel.png')
运行结果:
提取骨架之前的边界可视化结果:
提取的骨架的可视化结果:
用skimage.morphology中的skeletonize的函数提取的结果:
总结
- 提出的方法与传统的skeletonize(skimage)相比还是有一定的差距,特别是,从上面的可视化结果中,我们也可以看出来,该方法提取的估计存在很多断裂的情况,这对骨架来说是难以忍受的
- 该方法存在一个超参,即指定迭代的次数,这个参数也非常影响结果,上面的例子是使用的10,我试过5的情况,生成的骨架就更加槽糕了
- 但是该方法还是有一个最大的优势,就是它可微,可导,可以用于网络中输出骨架然后作为一项loss
边栏推荐
- Vs2019 initial use
- [analysis of teacher Gao's software needs] collection of exercises and answers for level 20 cloud class
- [exercise-5] (UVA 839) not so mobile (balance)
- 信息安全-史诗级漏洞Log4j的漏洞机理和防范措施
- Opencv learning log 24 -- Hough transform 2 (maximum interval and minimum length can be limited)
- The most complete programming language online API document
- JS call camera
- 渗透测试 ( 8 ) --- Burp Suite Pro 官方文档
- Borg Maze (BFS+最小生成树)(解题报告)
- mysql导入数据库报错 [Err] 1273 – Unknown collation: ‘utf8mb4_0900_ai_ci’
猜你喜欢
Frida hook so layer, protobuf data analysis
Penetration test (3) -- Metasploit framework (MSF)
Write web games in C language

Information security - Epic vulnerability log4j vulnerability mechanism and preventive measures
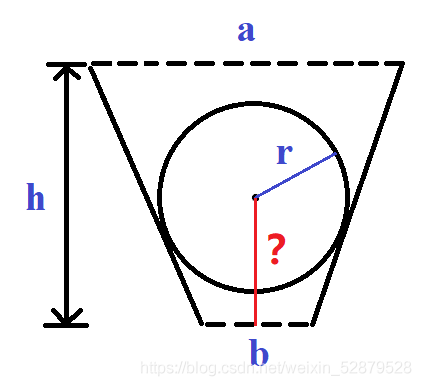
Ball Dropping
树莓派4B安装opencv3.4.0
1323. Maximum number of 6 and 9
Web based photo digital printing website
409. Longest palindrome
860. Lemonade change
随机推荐
滲透測試 ( 1 ) --- 必備 工具、導航
nodejs爬虫
信息安全-史诗级漏洞Log4j的漏洞机理和防范措施
1013. Divide the array into three parts equal to and
Ball Dropping
信息安全-威胁检测引擎-常见规则引擎底座性能比较
Research Report on surgical fluid treatment industry - market status analysis and development prospect prediction
Write web games in C language
X-Forwarded-For详解、如何获取到客户端IP
Gartner:关于零信任网络访问最佳实践的五个建议
921. Minimum additions to make parentheses valid
[exercise-8] (UVA 246) 10-20-30== simulation
7-1 懂的都懂 (20 分)
C language must memorize code Encyclopedia
【练习-11】4 Values whose Sum is 0(和为0的4个值)
[exercise-1] (UVA 673) parentheses balance/ balanced brackets (stack)
Nodejs+vue网上鲜花店销售信息系统express+mysql
Analyse du format protobuf du rideau en temps réel et du rideau historique de la station B
mysql导入数据库报错 [Err] 1273 – Unknown collation: ‘utf8mb4_0900_ai_ci’
Raspberry pie csi/usb camera uses mjpg to realize web camera monitoring