当前位置:网站首页>PyRosetta 安装方法之Conda安装
PyRosetta 安装方法之Conda安装
2022-08-02 20:32:00 【CDamogu】
1 作者说
在我下面的这篇文章中曾尝试过安装旧版本PyRosetta 3, 但是最后好像无疾而终.
Windows下Rosetta Commons ,PyRosetta软件安装方法,API在线及离线文档,Zeal工具安装方法
官方也提供了PyRosetta 4的安装方法,看着有点繁琐,这里提供一种比较合理的方法安装.
如果你该软件的基础知识,支持的平台,Rosetta和PyRosetta之间关系等有着迷茫的话,不妨先去 官方 FAQ ,磨刀不误砍柴工,真的是必备基础
2 前置条件
如果你和我一样,Windows11系统, Ubuntu安装过,那么推荐你使用下述方法
PyRosetta的安装可以通过:
- conda安装
- 源代码编译安装
两种方式都需要先在PyRosetta官网申请学术下载证书取得具有下载权限的用户名和密码。
3 Conda方式安装
2.1 conda安装
这里重点讲如何通过conda安装, 那么conda是什么? 暂且将其理解为包管理工具
官方考虑到没有root权限的用户的使用需求,给出了一条捷径,使用申请到的学术下载证书中的用户名和密码,可以建立一条特殊的conda channel,具体格式是: https://USERNAME:[email protected]
假如证书中的用户名和密码分别是CDamogu和abcdefg
那么你的conda channel就是https://CDamogu:[email protected]
但是具体什么时候用呢?后面会详细说明
- 安装conda
在Conda 官网 选择适合自己平台的版本右键复制下载链接并使用wget命令下载,运行安装包并根据提示安装,安装后还要激活才能使用,参考代码如下: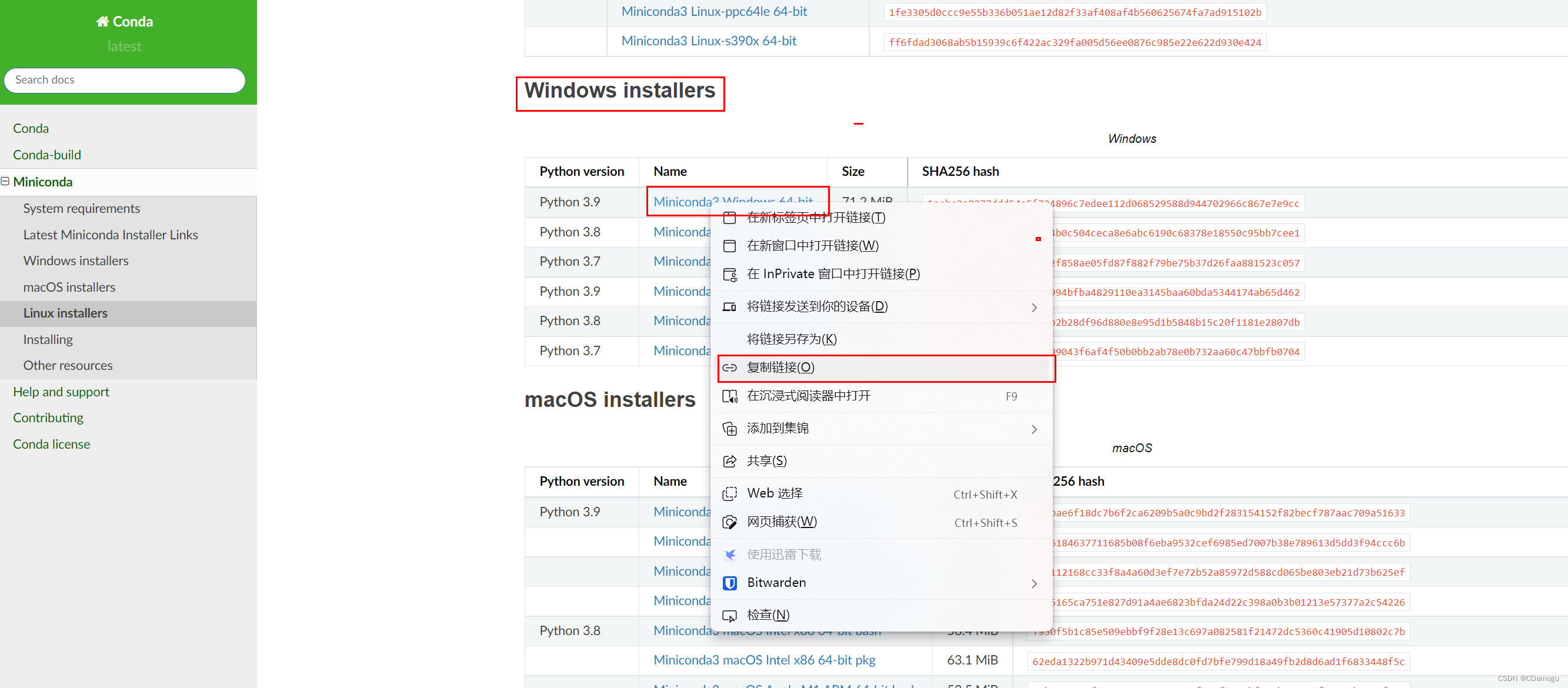
图片是拿windows距离,这里我们要装入Ubuntu,所以选择Linux的
# 下载
wget https://repo.anaconda.com/miniconda/Miniconda3-py39_4.12.0-Linux-x86_64.sh
# 安装
bash Miniconda3-latest-Linux-x86_64.sh
# 默认安装路径为家目录~
cd ~/miniconda3/bin
# #激活conda
source ./activate
如果对linux稍微有点基础的,会比较好,如果没有的话建议先看看cd,ls,等指令,下图是我已经安装好的
1.2. 激活与退出:
激活之后会看到命令提示符前出现了(base),说明已经在conda的默认环境中, 之后每次登陆服务器想要进入和退出conda环境,在conda环境中安装应用可通过以下代码实现: (这种感觉有点像Python的Venv环境的进入与退出)
# 进入conda环境
conda activate
# 离开conda环境
conda deactivate
# 安装XXX至conda环境
conda install XXX
1.3. 添加下载频道(镜像源):
conda的channel就是集成了各类应用的应用池,为conda用户提供搜索和下载,这里通过添加国内的镜像源提供稳定高速的conda使用,参考代码如下:
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
1.4 将 https://USERNAME:[email protected] 添加到.condarc的channel列表中,参考代码如下: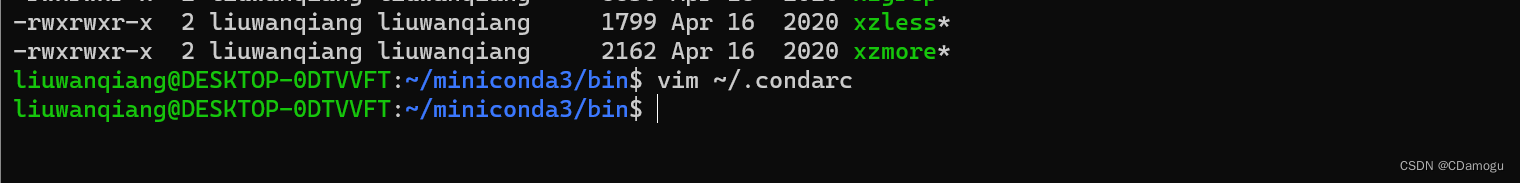
vim ~/.condarc
# 按 i 键进入输入模式,在下面列表中加入新的channel链接
# 新加入的下载channel
channels:
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
- https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
- https://CDamogu:[email protected]
# 按 Esc 键退出编辑模式,按 : 键,再输入wq,按 Enter 键保存退出
2.2. conda下载
PyRosetta下载和位于 ./miniconda3/pkgs/
# 安装最新的PyRosetta版本:
conda install pyrosetta
# 安装特定版本的PyRosetta:
conda install pyrosetta=<version>
3 PyRosetta使用方法
3.1 脚本参考
Structure Based Prediction of Neoantigen Immunogenicity
脚本内容如下, 具体实现的东西还是参考上述文献,目前也处于摸索阶段
''' peptide_MHC-modeling.py PyRosetta4 script for predictive structural modeling of nonameric peptide-MHC structures. Easily adaptable to other peptide lengths. Usage: python peptide_MHC-modeling.py template peptide n where template is the starting structure in PDB format peptide is the nonameric peptide to be modeled and n is the number of decoys to generate. It is assumed that the peptide is present in the template model as a separate chain, designated chain C. -- GLJ Keller and TP Riley '''
# stdlib imports
from __future__ import print_function
import os
from sys import argv
from random import randint
# PyRosetta4 initialization, enabling use of talaris scorefunctions
from pyrosetta import * ; from pyrosetta.rosetta import *
init(extra_options = "-extrachi_cutoff 12 -ex1 -ex2 -ex3 -corrections::restore_talaris_behavior")
# PyRosetta utility imports
from pyrosetta.toolbox import mutate_residue
_, template, peptide, n = argv
template_model = pose_from_pdb(template)
scorefxn = create_score_function('talaris2014')
positionlist = range(template_model.pdb_info().pdb2pose('C',1),template_model.pdb_info().pdb2pose('C',9)+1)
# create separate output directory for each peptide modeled
output_dir = '_models_{0}'.format(peptide)
if not os.path.exists(output_dir): os.mkdir(output_dir)
os.chdir(output_dir)
# PyRosetta job distributor to parallelize modeling
jd = PyJobDistributor(peptide, int(n), scorefxn)
jd.native_pose = template_model
while not jd.job_complete:
# assign starting model to new pose for later comparison
mutant = Pose() ; mutant.assign(template_model)
# mutate peptide residue in template structure at position i to res for each position in peptide
# (assumed chain C)
for i, res in enumerate(peptide):
mutate_residue(mutant, positionlist[i], res, 0.0, scorefxn)
# assign mutant model (with starting coordinates) to new pose for later comparison
remodel_target = Pose() ; remodel_target.assign(mutant)
# define loop peptide_ft to peptide residues p2 through p9, with the cutsite set randomly per decoy
peptide_ft = rosetta.protocols.loops.Loop(positionlist[1], positionlist[7], positionlist[randint(2, 6)])
peptide_loops = rosetta.protocols.loops.Loops() ; peptide_loops.add_loop(peptide_ft)
# define fold tree of model as peptide_ft object
rosetta.protocols.loops.set_single_loop_fold_tree(remodel_target, peptide_ft)
# create repacking task; disallow changing residue identity; allow repacking to current rotamer
task_pack = rosetta.core.pack.task.TaskFactory.create_packer_task(remodel_target)
task_pack.restrict_to_repacking() ; task_pack.or_include_current(True)
pack = rosetta.protocols.minimization_packing.PackRotamersMover(scorefxn, task_pack)
# repack model after mutation
pack.apply(remodel_target)
# use Cyclic Coordinate Descent algorithm to remodel peptide coordinates with flexible backbone
loops_refine_CCD = rosetta.protocols.loops.loop_mover.refine.LoopMover_Refine_CCD(peptide_loops, scorefxn)
loops_refine_CCD.max_inner_cycles(10)
loops_refine_CCD.apply(remodel_target)
# finally, repack and output final model
pack.apply(remodel_target)
jd.output_decoy(remodel_target)
3.2 使用方式
下载脚本.py后,将其放入 ./miniconda3/pkgs/pyrosetta/lib/python/site-packages/ 路径下,启动conda环境通过python调用,在我这大概4分钟完成一个结构的建模,输出.pdb结构文件和一个包含物化信息的文本.
3.3 建议
- 具体路径名根据下载版本不同可能存在差异,请根据具体情况修改
- 面对大批量的建模任务时建议编写循环脚本配合OS模块实现自动化
边栏推荐
- Mysql用户管理
- 汉源高科2光12电千兆导轨式网管型工业以太网交换机双光自愈保护式以太网光交换机
- 李沐动手学深度学习V2-bert和代码实现
- 信息学奥赛一本通(1257:Knight Moves)
- 美国爱荷华州立大学| Improving Distantly Supervised Relation Extraction by Natural Language Inference(通过自然语言推理改进远程监督关系提取)
- setup语法糖 defineProps defineEmits defineExpose
- Tencent YunMeng every jie: I experienced by cloud native authors efficiency best practices case
- 典型相关分析CCA计算过程
- 如何使用windbg查看C#某个线程的栈大小 ?
- C# Barrier类
猜你喜欢

【数据分析】:什么是数据分析?

交 叉 数 组

Digital twins help visualize the construction of smart cities

Flink Yarn Per Job - 创建启动Dispatcher RM JobManager
A brief discussion on the transformation of .NET legacy applications
Jar包启动通过ClassPathResource获取不到文件路径问题
OP-5,输入/输出信号范围-一信号处理能力
模糊查询like用法实例(Bee)
DataGrip 安装教程 详细版
How to quickly compare two byte arrays for equality in .NET
随机推荐
The time series database has been developed for 5 years. What problem does it need to solve?
C# Monitor class
「每周译Go」这次我们来点不一样的!--《How to Code in Go》系列上线
汉源高科千兆4光4电工业级网管型智能环网冗余以太网交换机防浪涌防雷导轨式安装
千人优学 | GBase 8s数据库2022年6月大学生专场实训圆满结束
The Orsay in Informatics (1256: Bouquet for Algernon)
Flink Yarn Per Job - 启动AM
回文自动机+CodeTON Round 2 C,D
信息学奥赛一本通(1257:Knight Moves)
KDD 2022 | 深度图神经网络中的特征过相关:一个新视角
ICLR 2022最佳论文:基于对比消歧的偏标签学习
Bena's life cycle
二叉搜索树的实现
Li Mu hands-on learning deep learning V2-bert and code implementation
你所不知道的C#中的细节
php 单引号 双引号 -> => return echo
2022年金九银十,Android面试中高频必问的问题汇总
「 每日一练,快乐水题 」1374. 生成每种字符都是奇数个的字符串
Helm基础知识
Informatics Olympiad All-in-One (1257: Knight Moves)