当前位置:网站首页>tidyverse based on data.table?
tidyverse based on data.table?
2022-08-03 20:42:00 【A Yue 1229】
获取更多R语言知识,请关注公众号:医学和生信笔记
“医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化.主要分享R语言做医学统计学、meta分析、网络药理学、临床预测模型、机器学习、生物信息学等.
tidyverse作为RThe Swiss Army Knife in Linguistic Data Analysis,非常好用,A small downside is that it is slow,data.table速度快,So their team developed it againdtplyr,加快运行速度.
But today is another one,基于data.table的tidyverse:tidytable.
使用起来非常简单,Just add one after the original function.即可!!!
Below is a simple speed comparison of a common operation,It can be seen that the speed has been greatly improved~
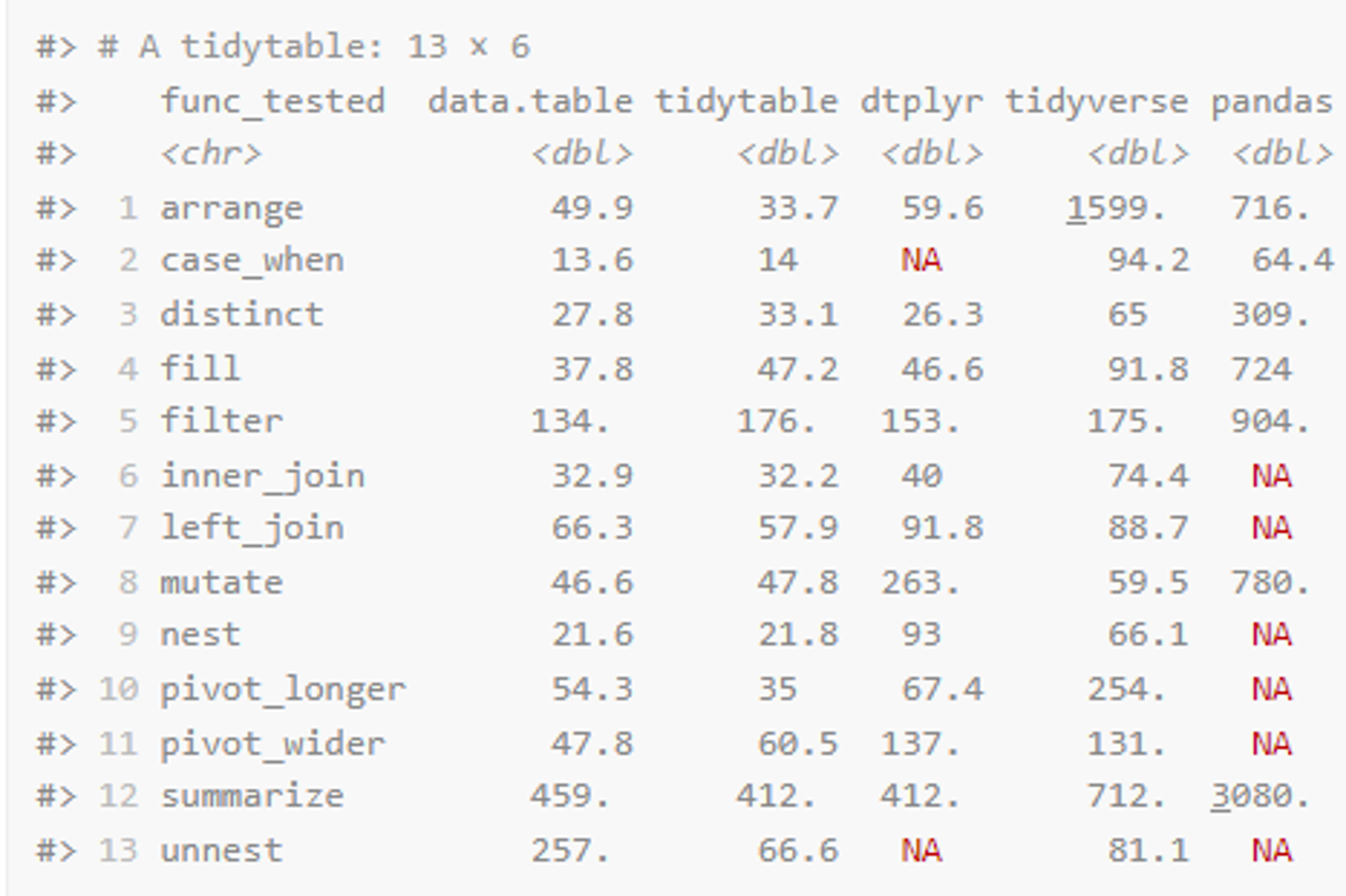
安装
# 经典2选1
install.packages("tidytable")
# install.packages("devtools")
devtools::install_github("markfairbanks/tidytable")
一般使用
Just add one after the function.就可以了!!
library(tidytable)
## Warning: package 'tidytable' was built under R version 4.2.1
##
## Attaching package: 'tidytable'
## The following object is masked from 'package:stats':
##
## dt
df <- data.table(x = 1:3, y = 4:6, z = c("a", "a", "b"))
df %>%
select.(x, y, z) %>%
filter.(x < 4, y > 1) %>%
arrange.(x, y) %>%
mutate.(double_x = x * 2,
x_plus_y = x + y)
## # A tidytable: 3 × 5
## x y z double_x x_plus_y
## <int> <int> <chr> <dbl> <int>
## 1 1 4 a 2 5
## 2 2 5 a 4 7
## 3 3 6 b 6 9
分组汇总
和group_by()稍有不同,这里需要使用.by = 进行分组汇总.
df %>%
summarize.(avg_x = mean(x),
count = n(),
.by = z) # Grouping summary forms are different
## # A tidytable: 2 × 3
## z avg_x count
## <chr> <dbl> <int>
## 1 a 1.5 2
## 2 b 3 1
每次都要调用:
df <- data.table(x = c("a", "a", "a", "b", "b"))
df %>%
slice.(1:2, .by = x) %>% # .by
mutate.(group_row_num = row_number(), .by = x) # .by
## # A tidytable: 4 × 2
## x group_row_num
## <chr> <int>
## 1 a 1
## 2 a 2
## 3 b 1
## 4 b 2
支持tidyselect
常见的everything(), starts_with(), ends_with(), any_of(), where()等都是支持的.
df <- data.table(
a = 1:3,
b1 = 4:6,
b2 = 7:9,
c = c("a", "a", "b")
)
df %>%
select.(a, starts_with("b"))
## # A tidytable: 3 × 3
## a b1 b2
## <int> <int> <int>
## 1 1 4 7
## 2 2 5 8
## 3 3 6 9
df %>%
select.(-a, -starts_with("b"))
## # A tidytable: 3 × 1
## c
## <chr>
## 1 a
## 2 a
## 3 b
可以和.by连用:
df <- data.table(
a = 1:3,
b = c("a", "a", "b"),
c = c("a", "a", "b")
)
df %>%
summarize.(avg_a = mean(a),
.by = where(is.character))
## # A tidytable: 2 × 3
## b c avg_a
## <chr> <chr> <dbl>
## 1 a a 1.5
## 2 b b 3
支持data.table语法
借助dt()函数实现对data.table语法的支持.
df <- data.table(x = 1:3, y = 4:6, z = c("a", "a", "b"))
df %>%
dt(, .(x, y, z)) %>%
dt(x < 4 & y > 1) %>%
dt(order(x, y)) %>%
dt(, double_x := x * 2) %>%
dt(, .(avg_x = mean(x)), by = z)
## # A tidytable: 2 × 2
## z avg_x
## <chr> <dbl>
## 1 a 1.5
## 2 b 3
基本上tidyverseAll functions related to data analysis can be used,A detailed list of supported functions can be found here这里[1]找到.
获取更多R语言知识,请关注公众号:医学和生信笔记
“医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化.主要分享R语言做医学统计学、meta分析、网络药理学、临床预测模型、机器学习、生物信息学等.
参考资料
tidytable支持的函数: https://markfairbanks.github.io/tidytable/reference/index.html
边栏推荐
- ES6 - Arrow Functions
- 关于shell脚本的一些思考
- leetcode 16.01. Swap numbers (swap the values of 2 numbers without using temporary variables)
- Go语言类型与接口的关系
- ESP8266-Arduino编程实例-BH1750FVI环境光传感器驱动
- 云服务器如何安全使用本地的AD/LDAP?
- 2022年1~7月语音合成(TTS)和语音识别(ASR)论文月报
- 倒计时2天,“文化数字化战略新型基础设施暨文化艺术链生态建设发布会”启幕在即
- 肝完 Alibaba 这份面试通关宝典,我成功拿下今年第 15 个 Offer
- 5 款漏洞扫描工具:实用、强力、全面(含开源)
猜你喜欢
随机推荐
Why BI software can't handle correlation analysis
RNA核糖核酸修饰Alexa 568/[email protected] 594/[email prote
后台图库上传功能
直播源码开发,各种常见的广告形式
Markdown语法
详解虚拟机!京东大佬出品 HotSpot VM 源码剖析笔记(附完整源码)
How can a cloud server safely use local AD/LDAP?
AWTK开发编译环境踩坑记录1(编译提示powershell.exe出错)
15 years experience in software architect summary: in the field of ML, tread beginners, five hole
leetcode 136. 只出现一次的数字(异或!!)
华为设备配置VRRP与BFD联动实现快速切换
Power button 206 - reverse list - the list
双线性插值公式推导及Matlab实现
刷题错题录1-隐式转换与精度丢失
Likou 707 - Design Linked List - Linked List
Go语言为任意类型添加方法
canvas螺旋动画js特效
ARMuseum
leetcode 136. Numbers that appear only once (XOR!!)
从开发到软件测试:除了扎实的测试基础,还有哪些必须掌握 ?