当前位置:网站首页>A Competitive Swarm Optimizer for Large Scale Optimization
A Competitive Swarm Optimizer for Large Scale Optimization
2022-07-27 05:46:00 【身影王座】
0、论文背景
本文实在PSO的基础上创新算法,得到新的算法CSO。在提出的CSO中,每个粒子的个人最佳位置和全局最佳位置(或邻域最佳位置)都不参与粒子的更新。相反,引入了两两竞争机制,失去竞争的粒子将通过向获胜者学习来更新其位置。
Cheng R, Jin Y. A competitive swarm optimizer for large scale optimization[J]. IEEE transactions on cybernetics, 2014, 45(2): 191-204.

1、CSO
PSO算法之前有讲过,参见博客:PSO。
然而,我们发现,当优化问题具有大量的局部优化或为高维时,PSO表现较差。上述弱点通常可以归因于在PSO中经常发生的过早收敛。在实践中,由于gbest的全球影响,pbest很可能具有与gbest相似甚至相同的值,从而大大减少了蜂群的分化。
在这项工作中,我们探索了单个群内粒子之间的竞争机制的使用。此外,输掉比赛的粒子将向获胜的粒子学习,而不是向gbest或pbesti学习。
1.1 竞争机制
在每一代中,P(t)中的粒子被随机分配到m/2对(假设群大小m是偶数),然后在每对中的两个粒子之间进行竞争。作为每一场比赛的结果,适应性更好的粒子,以下称为获胜者,将直接传递给下一代的群体P(t+1)。而失去竞争的粒子(失败者),将通过学习从赢家更新其位置和速度。在向获胜者学习后,失败者也将被传递给蜂群P(t+1)。

1.2 粒子位置更新机制


 是相关粒子的平均位置值,
是相关粒子的平均位置值, 是控制
是控制 影响的参数。具体来说,对于
影响的参数。具体来说,对于 ,可以采用全局版本和本地版本:
,可以采用全局版本和本地版本:
 为P(t)中所有粒子的全局平均位置。
为P(t)中所有粒子的全局平均位置。 表示粒子在粒子l的预定义邻域中的局部平均位置。
表示粒子在粒子l的预定义邻域中的局部平均位置。
如果采用PSO中朝gbest和pbest方向更新的话,会存在下面陷入局部最优的问题:

如果在PSO中去掉gbest,只朝pbest方向更新的话,会有两种情况,同时一种会存在下面陷入局部最优的问题:


而利用竞争机制,随机选两个粒子,进行二者之间相互竞争学习来更新,就会有效避免陷入上述局部最优的问题:

1.3 算法流程

2、算法复现以及简单实验
这里主要比较标准PSO与CSO,实验采用了一个测函数,函数评估次数保持一致,最后重复实验100次,取平均值。
clc;clear;clearvars;
% 随机生成10个数据
num_initial = 10;
num_vari = 30;
% 搜索区间
upper_bound = 5.12;
lower_bound = -5.12;
iter = 20000;
varphi = 1.5;
% 随机生成10个数据,并获得其评估值
sample_x = lhsdesign(num_initial, num_vari).*(upper_bound - lower_bound) + lower_bound.*ones(num_initial, num_vari);
sample_y = Rastrigin(sample_x);
Fmin = zeros(iter, 1);
aver_Fmin = zeros(iter, 1);
for n = 1 : 100
k = 1;
% 初始化一些参数
X = sample_x;
Y = sample_y;
V = sample_x;
[fmin, ~] = min(Y);
fprintf("n: %.4f\n", n);
UV = V;
UX = X;
UY = Y;
for i = 1 : iter
PV = [];
PX = [];
PY = [];
while ~isempty(UX)
m = size(UX, 1);
k1 = randi(m);
k2 = randi(m);
while k2 == k1
k2 = randi(m);
end
v1 = UV(k1, :);
x1 = UX(k1, :);
y1 = UY(k1, :);
v2 = UV(k2, :);
x2 = UX(k2, :);
y2 = UY(k2, :);
r1 = rand(1, num_vari);
r2 = rand(1, num_vari);
r3 = rand(1, num_vari);
if y1 <= y2
PV = [PV; v1];
PX = [PX; x1];
PY = [PY; y1];
v2 = r1 .* v2 + r2 .* (x1 - x2) + varphi * r3 .* ((x1 + x2) ./ 2 - x2);
x2 = x2 + v2;
x2(x2 > upper_bound) = upper_bound;
x2(x2 < lower_bound) = lower_bound;
y2 = Rastrigin(x2);
PV = [PV; v2];
PX = [PX; x2];
PY = [PY; y2];
else
PV = [PV; v2];
PX = [PX; x2];
PY = [PY; y2];
v1 = r1 .* v1 + r2 .* (x2 - x1) + varphi * r3 .* ((x1 + x2) ./ 2 - x1);
v1(v1 > upper_bound) = upper_bound;
v1(v1 < lower_bound) = lower_bound;
x1 = x1 + v1;
x1(x1 > upper_bound) = upper_bound;
x1(x1 < lower_bound) = lower_bound;
y1 = Rastrigin(x1);
PV = [PV; v1];
PX = [PX; x1];
PY = [PY; y1];
end
if k1 > k2
UV(k1, :) = [];
UX(k1, :) = [];
UY(k1, :) = [];
UV(k2, :) = [];
UX(k2, :) = [];
UY(k2, :) = [];
else
UV(k2, :) = [];
UX(k2, :) = [];
UY(k2, :) = [];
UV(k1, :) = [];
UX(k1, :) = [];
UY(k1, :) = [];
end
end
UV = PV;
UX = PX;
UY = PY;
% 更新所有个体最佳位置
[fmin, ~] = min(UY);
% fprintf("iter %d fmin: %.4f\n", i, fmin);
Fmin(k, 1) = fmin;
k = k +1;
end
aver_Fmin = aver_Fmin + Fmin;
end
aver_Fmin = aver_Fmin ./ 100;
% disp(pbestx(gbest, :));
plot(aver_Fmin);PSO结果:

CSO(varphi = 0.1)时:

CSO(varphi = 0.5)时:

CSO(varphi = 1)时:

CSO(varphi = 1.5)时:

从目前结果来看,CSO效果远远好于PSO,而且收敛速度也远远快于PSO。同时我们也注意到varphi=1时候效果最好,但是varphi越大或者越小收敛速度也就越快,但是搜索的效果也有可能会下降。
边栏推荐
- Web configuration software for industrial control is more efficient than configuration software
- ZnS-DNA QDs近红外硫化锌ZnS量子点改性脱氧核糖核酸DNA|DNA修饰ZnS量子点
- PHP defines the array using commas,
- PNA polypeptide PNA TPP | GLT ala ala Pro Leu PNA | suc ala Pro PNA | suc AAPL PNA | suc AAPM PNA
- deepsort源码解读(一)
- Gbase 8C - SQL reference 6 SQL syntax (15)
- pre-commit install 时 CalledProcessError
- Leetcode series (I): buying and selling stocks
- TS learning (VIII): classes in TS
- deepsort源码解读(七)
猜你喜欢

火狐浏览器,访问腾讯云服务器的时候,出现建立安全连接失败的问题。
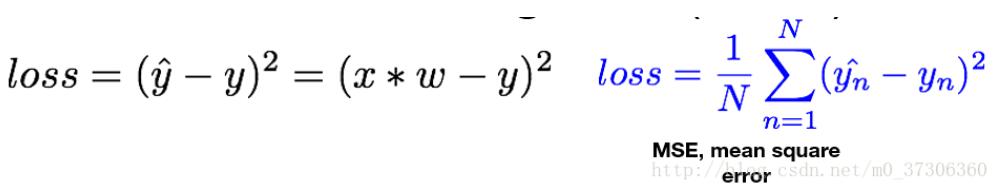
Reflection on pytorch back propagation

Talk about multimodality of fire

DNA(脱氧核糖核酸)供应|碳纳米管载核酸-DNA/RNA材料|DNA/RNA核酸修饰磁性纳米颗粒

How to make the minimum API bind the array in the query string

ZnS DNA QDs near infrared zinc sulfide ZnS quantum dots modified deoxyribonucleic acid dna|dna modified ZnS quantum dots
Dimension problems and contour lines

PNA modified polypeptide arms PNA PNA DNA suc aapf PNA suc - (ALA) 3 PNA

pytorch笔记:TD3

PNA修饰多肽ARMS-PNA|PNA-DNA|suc-AAPF-pNA|Suc-(Ala)3-pNA
随机推荐
CASS11.0.0.4 for AutoCAD2010-2023免狗使用方法
Quartus:往别人的工程添加.v文件报错
Student achievement management system based on SSM
Interpretation of deepsort source code (I)
Peptide nucleic acid oligomer containing azobenzene monomer (nh2-tnt4, n-pnas) Qiyue biological customization
Music website management system based on SSM
VIVO应用市场APP上架总结
Analysis of pix2pix principle
基于SSM音乐网站管理系统
New features of ES6 (2)
What is OKR and what is the difference between OKR and KPI
齐岳:巯基修饰寡聚DNA|DNA修饰CdTe/CdS核壳量子点|DNA偶联砷化铟InAs量子点InAs-DNA QDs
AI: play games in your spare time - earn it a small goal - [Alibaba security × ICDM 2022] large scale e-commerce map of risk commodity inspection competition
TS learning (VIII): classes in TS
Interpretation of deepsort source code (III)
网易云信亮相 GIAC 全球互联网架构大会,解密新一代音视频架构在元宇宙场景的实践...
内部类--看这篇就懂啦~
Consideration on how the covariance of Kalman filter affects the tracking effect of deepsort
ZnS-DNA QDs近红外硫化锌ZnS量子点改性脱氧核糖核酸DNA|DNA修饰ZnS量子点
CdS quantum dots modified DNA | CDs DNA QDs | near infrared CdS quantum dots coupled DNA specification information
 为P(t)中所有粒子的全局平均位置。
为P(t)中所有粒子的全局平均位置。 表示粒子在粒子l的预定义邻域中的局部平均位置。
表示粒子在粒子l的预定义邻域中的局部平均位置。