当前位置:网站首页>【练一下1】糖尿病遗传风险检测挑战赛 【讯飞开放平台】
【练一下1】糖尿病遗传风险检测挑战赛 【讯飞开放平台】
2022-08-05 05:12:00 【墨苏玩电脑】
文章目录
完成coggle任务
任务1:报名比赛
步骤1:报名比赛http://challenge.xfyun.cn/topic/info?type=diabetes&ch=ds22-dw-zmt05
赛事
在这次比赛中,您需要通过训练数据集构建糖尿病遗传风险预测模型,然后预测出测试数据集中个体是否患有糖尿病。对于测试数据集当中的个体,您必须预测其是否患有糖尿病(患有糖尿病:1,未患有糖尿病:0),预测值只能是整数1或者0。
训练集(比赛训练集.csv)一共有5070条数据,用于构建您的预测模型(您可能需要先进行数据分析)。数据的字段有编号、性别、出生年份、体重指数、糖尿病家族史、舒张压、口服耐糖量测试、胰岛素释放实验、肱三头肌皮褶厚度、患有糖尿病标识(最后一列),您也可以通过特征工程技术构建新的特征。
测试集(比赛测试集.csv)一共有1000条数据,用于验证预测模型的性能。数据的字段有编号、性别、出生年份、体重指数、糖尿病家族史、舒张压、口服耐糖量测试、胰岛素释放实验、肱三头肌皮褶厚度。
提交说明
对于测试数据集当中的个体,您必须预测其是否患有糖尿病(患有糖尿病:1,未患有糖尿病:0),预测值只能是整数1或者0。提交的数据应该具有如下格式:
uuid,label
1,0
2,1
3,1
…
本次比赛中,预测模型的结果文件需要命名成:预测结果.csv,然后提交。请确保您提交的文件格式规范。
步骤2:下载比赛数据(点击比赛页面的赛题数据)
字段说明
编号:标识个体身份的数字;
性别:1表示男性,0表示女性;
出生年份:出生的年份;
体重指数:体重除以身高的平方,单位kg/m2;
糖尿病家族史:标识糖尿病的遗传特性,记录家族里面患有糖尿病的家属,分成三种标识,分别是父母有一方患有糖尿病、叔叔或者姑姑有一方患有糖尿病、无记录;
舒张压:心脏舒张时,动脉血管弹性回缩时,产生的压力称为舒张压,单位mmHg;
口服耐糖量测试:诊断糖尿病的一种实验室检查方法。比赛数据采用120分钟耐糖测试后的血糖值,单位mmol/L;
胰岛素释放实验:空腹时定量口服葡萄糖刺激胰岛β细胞释放胰岛素。比赛数据采用服糖后120分钟的血浆胰岛素水平,单位pmol/L;
肱三头肌皮褶厚度:在右上臂后面肩峰与鹰嘴连线的重点处,夹取与上肢长轴平行的皮褶,纵向测量,单位cm;
患有糖尿病标识:数据标签,1表示患有糖尿病,0表示未患有糖尿病。
步骤3:解压比赛数据,并使用pandas进行读取;
import pandas as pd
train_df = pd.read_csv("./比赛训练集.csv", encoding='gbk')
test_df = pd.read_csv("./比赛测试集.csv", encoding='gbk')
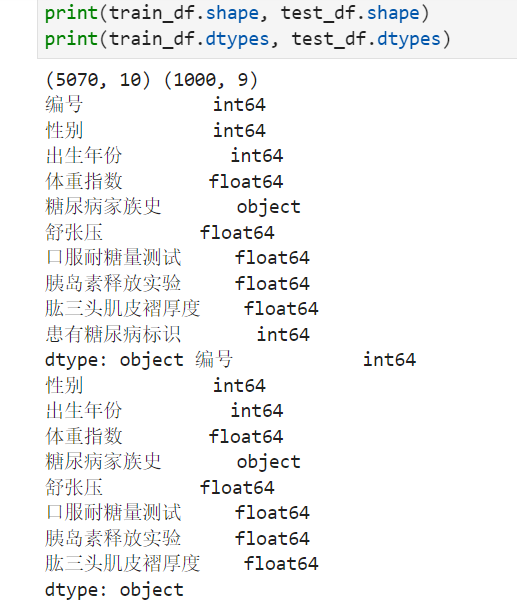
print(train_df.shape, test_df.shape)
print(train_df.dtypes, test_df.dtypes)
步骤4:查看训练集和测试集字段类型,并将数据读取代码写到博客;
任务2:比赛数据分析
步骤1:统计字段的缺失值,计算缺失比例;
通过缺失值统计,训练集和测试集的缺失值分布是否一致?
通过缺失值统计,有没有缺失比例很高的列?
#缺失值计算
train_df.isnull().mean(0)
test_df.isnull().mean(0)
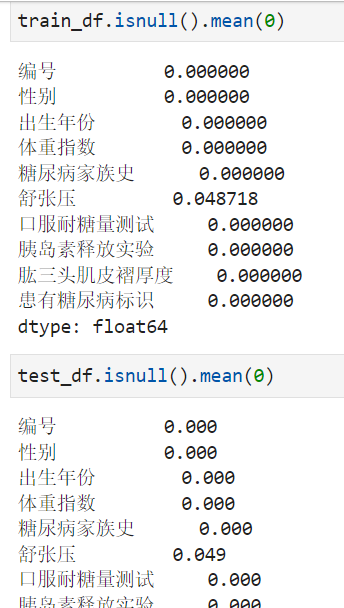
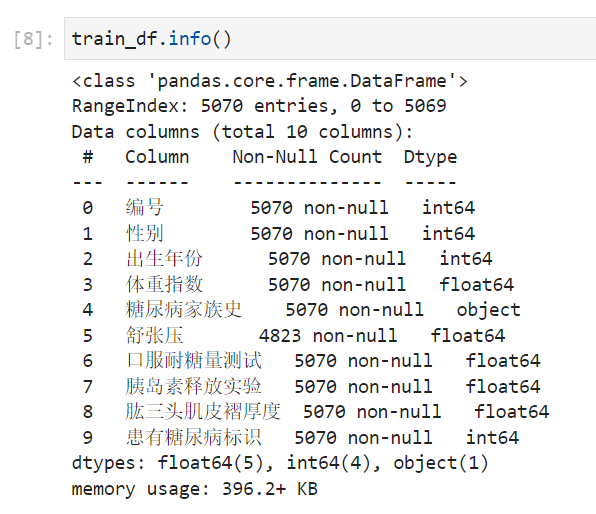
可以看出舒张压这个字段缺失了 4.9%。测试集和训练集比例差不多的。
步骤2:分析字段的类型;
从info和head(查看前5行的数据)可以看出,体重,舒张压,口服耐糖量测试 ,胰岛素释放实验,肱三头肌皮褶厚度是连续型,其余都是整型(糖尿病标识是0 1)。家族史那一列是备注(后期这个特征应该要由字符型可以处理为整型加入模型的使用)。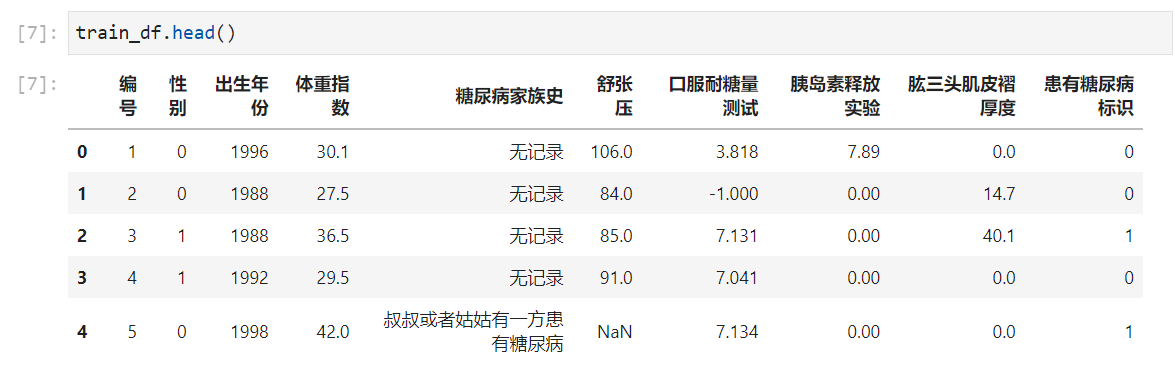
步骤3:计算字段相关性;
通过.corr()计算字段之间的相关性;
# 相关性计算
train_df.corr()
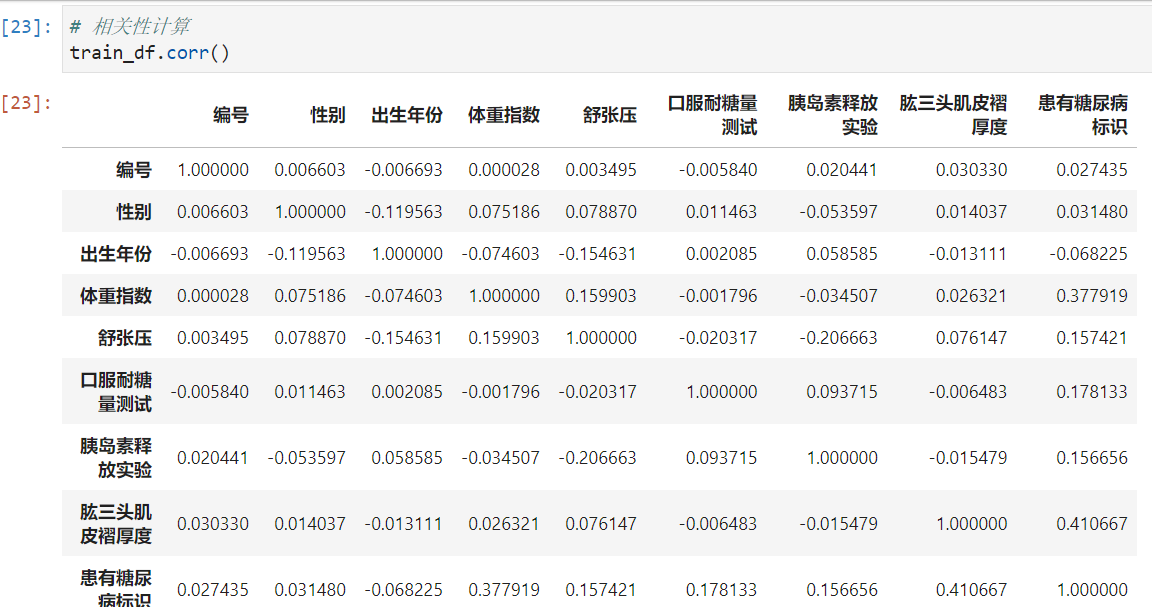
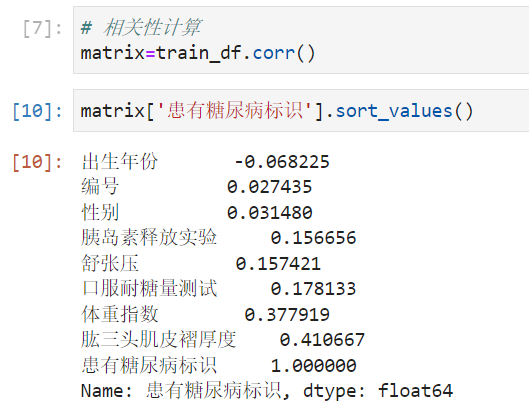
哪些字段与标签的相关性最高
- 可以看出 肱三头肌皮褶厚度和体重系数和糖尿病关系比较大。
可视化
尝试使用其他可视化方法将字段 与 标签的分布差异进行可视化;
以下也是用图表看一下糖尿病之间的分布。
之前没用过seaborn,boxplot应该是箱式图,countplot,和violinplot就不知道了
查了一下,countplot就是直方图,violinplot是小提琴样式的箱线图。
出现上图的原因是1.seaborn的几个图都叠在一起了,2.我语言设置没有切换为中文也没用(之后加入设置代码,这样中文和负坐标轴的符号才能正常显示),从老师那里偷的图片设置svg语句,更清楚一些
import seaborn as sns
import matplotlib.pyplot as plt
plt.rcParams['font.family'] = ['SimHei'] #用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False #用来正常显示负号
from IPython.display import set_matplotlib_formats
set_matplotlib_formats('svg')#展示图片格式
# train_df['性别'].value_counts().plot(kind='barh')# barh水平的,kind类型
# sns.countplot(x='患有糖尿病标识', hue='性别', data=train_df)#hue分列 value_counts和 countplot其实是一样的
# sns.boxplot(y='出生年份', x='患有糖尿病标识', hue='性别', data=train_df)
sns.violinplot(y="体重指数", x="患有糖尿病标识", hue="性别", data=train_df)
依次注释图像语句,得到下列图片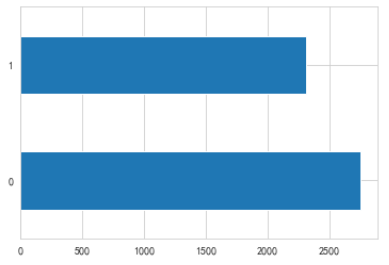
1是男性,0是女性。女性数据多一些
barh 水平的
横坐标轴0是没得糖尿病的,蓝色竖条0是女性。反正都是女性多一些,这也看不出糖尿病和性别的相关关系。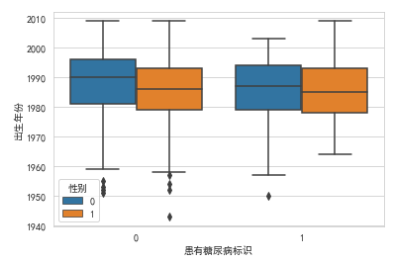
男性黄条的都算正态分布吧,女性蓝条的有点上移,不怎么正态。没得糖尿病的男的,有几个很长寿的点(离群点)。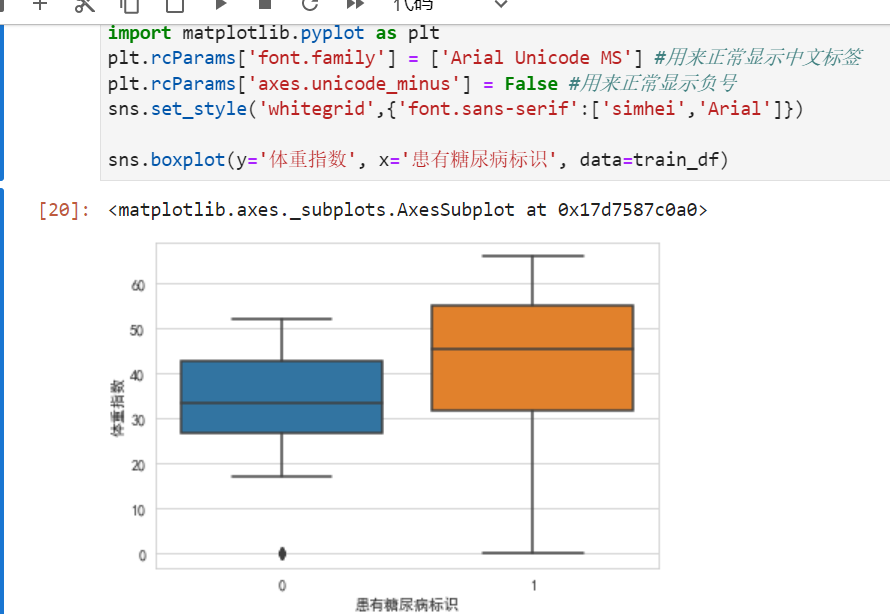
糖尿病人的体重分布比正常的要偏上一些。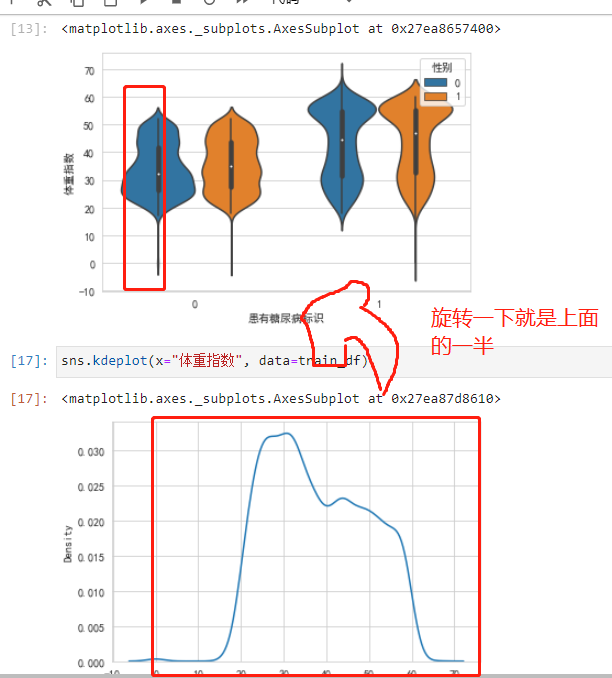
小提琴图其实就是测过来的密度直方图,对称的,看一半就好了.
scatterplot 散点图,xy都是数值型
然后 每个图都有一些 属性,像什么填充fill 还有 alpha透明度,自己可以去官网学。
tips:根据xy轴的数据类型来定用什么图
根据这些来判断能不能 做交叉特征(两列的加减乘除),能不能分离
补充学习 b站7.1录播
groupby
apply
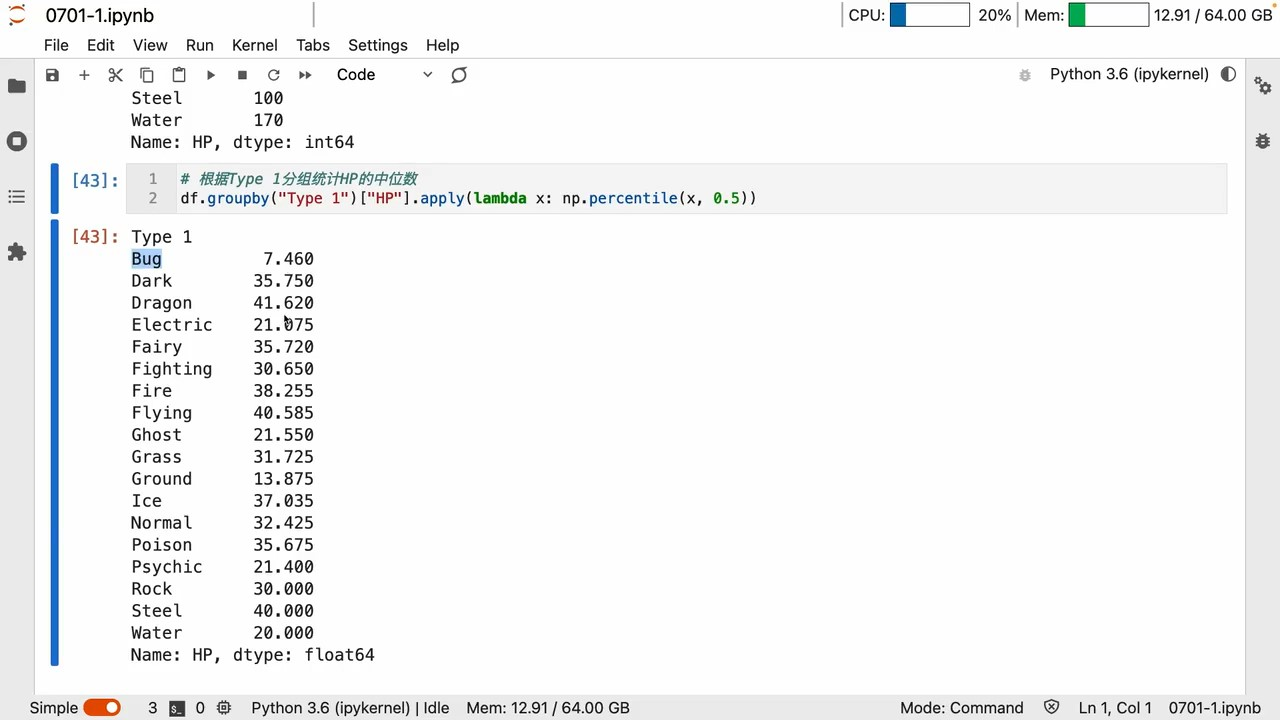
apply用 lambda 可以自定义许多
agg
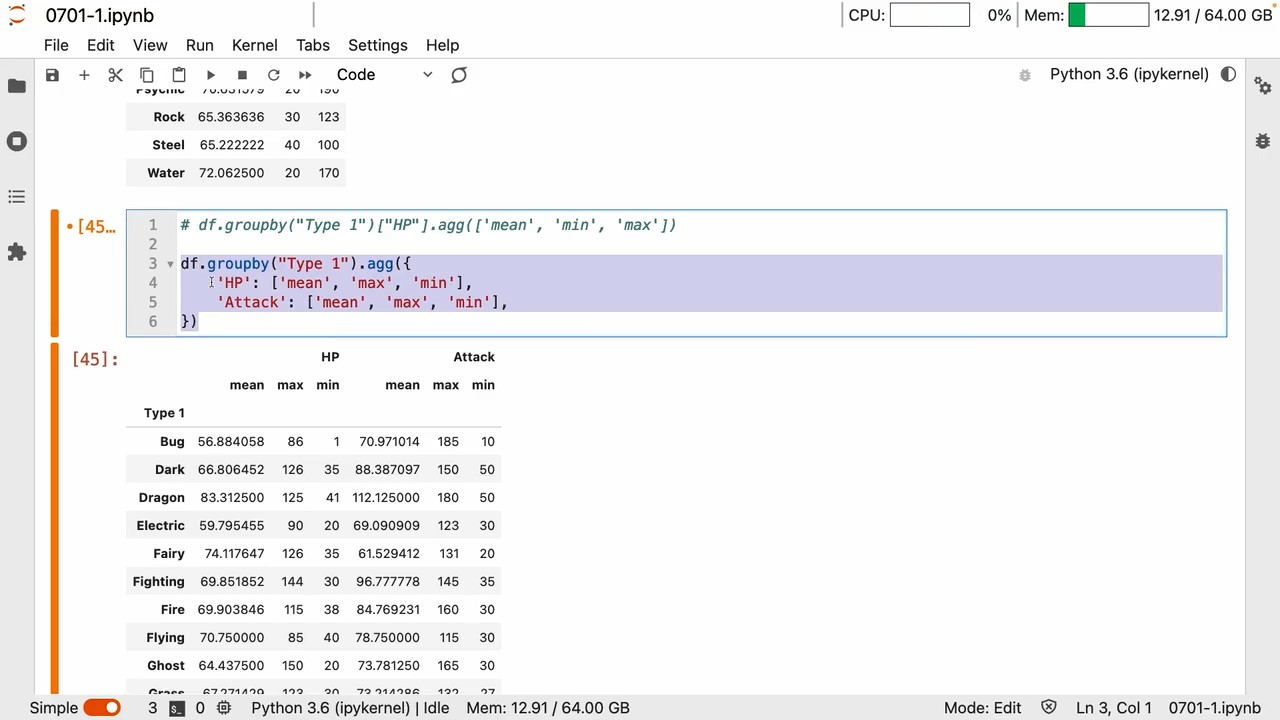
- groupby 和 agg 可以实现优雅的聚合效果
map、transform
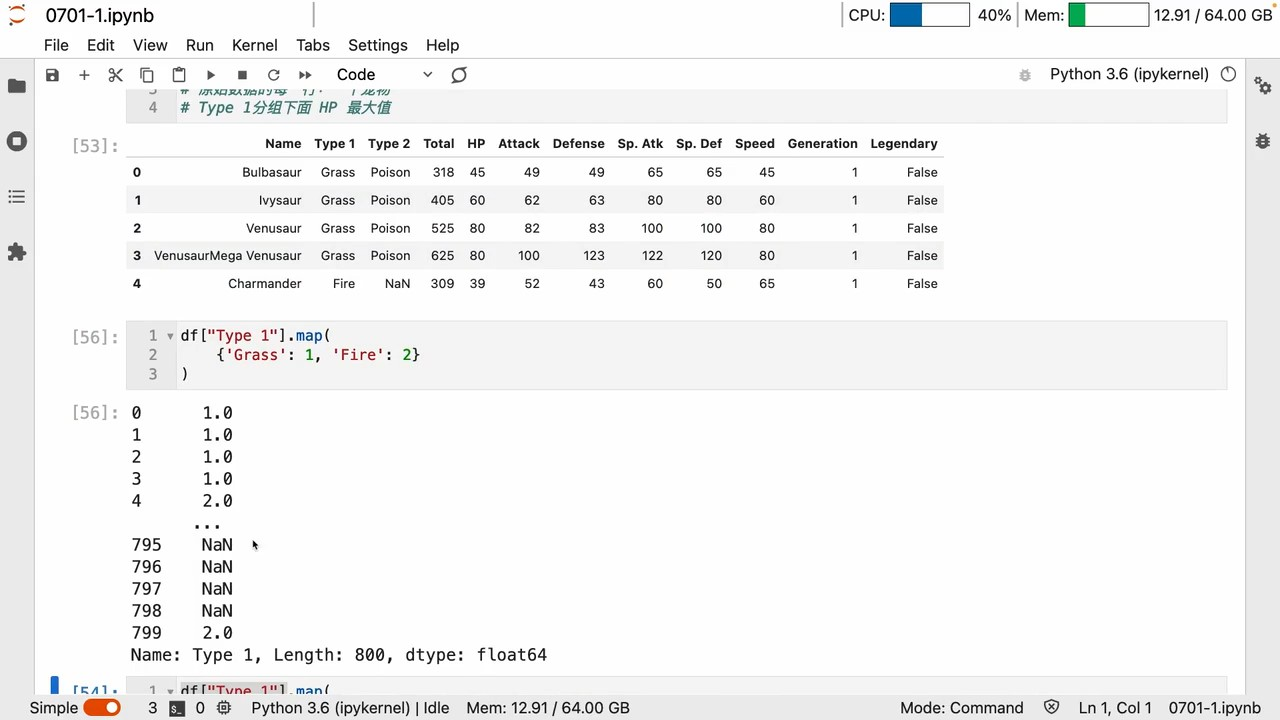 map可以映射改变 键值
map可以映射改变 键值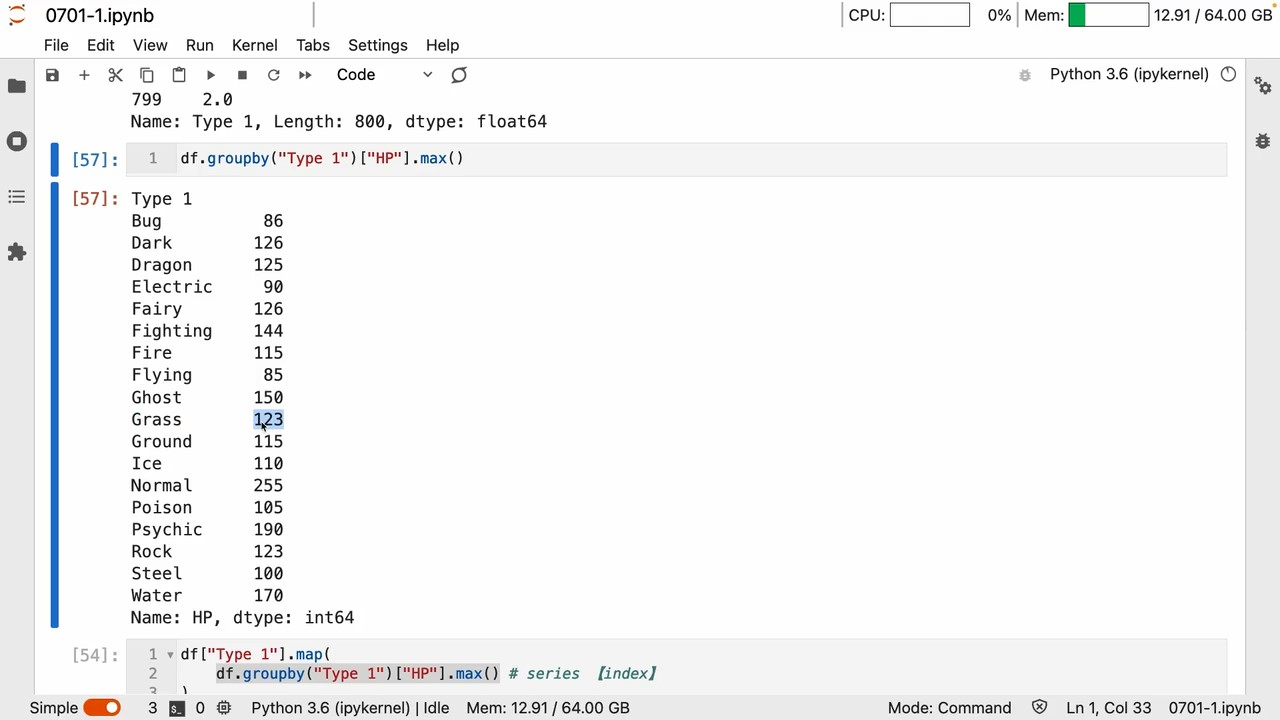
groupby 一系列操作又是得到键值关系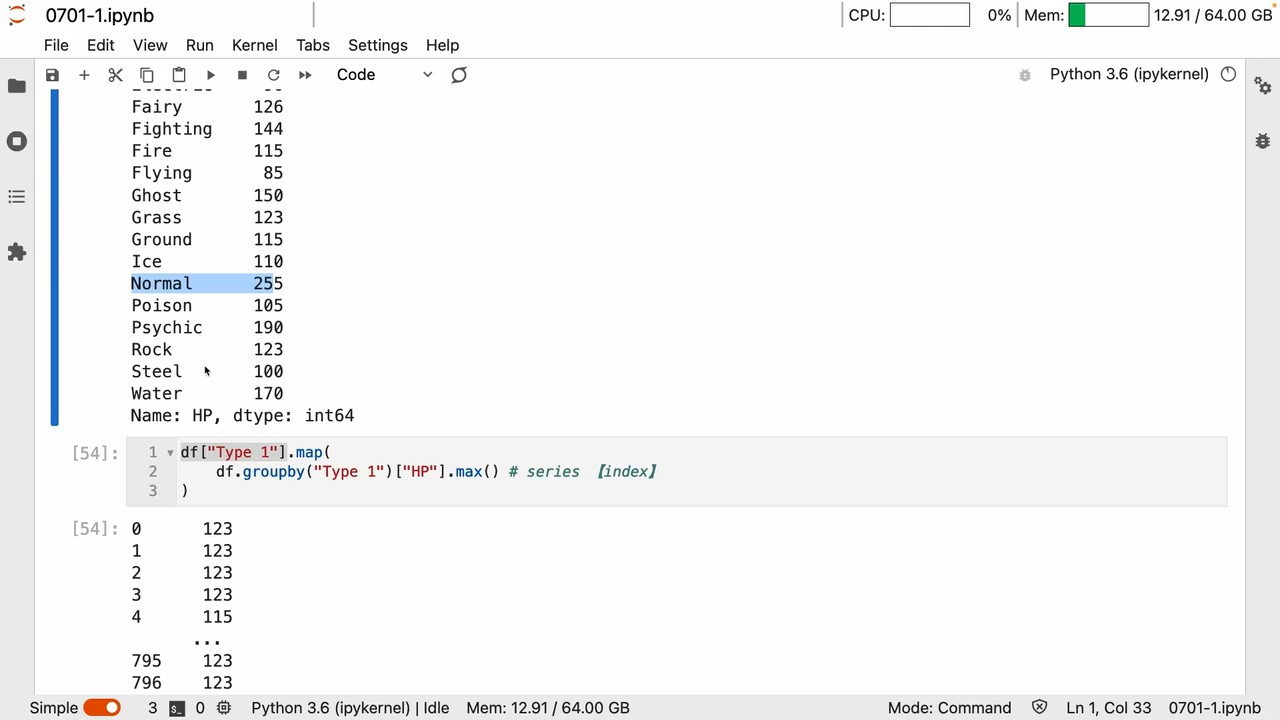
所以 map里面写个groupby 自然而然就可以映射替换了
实例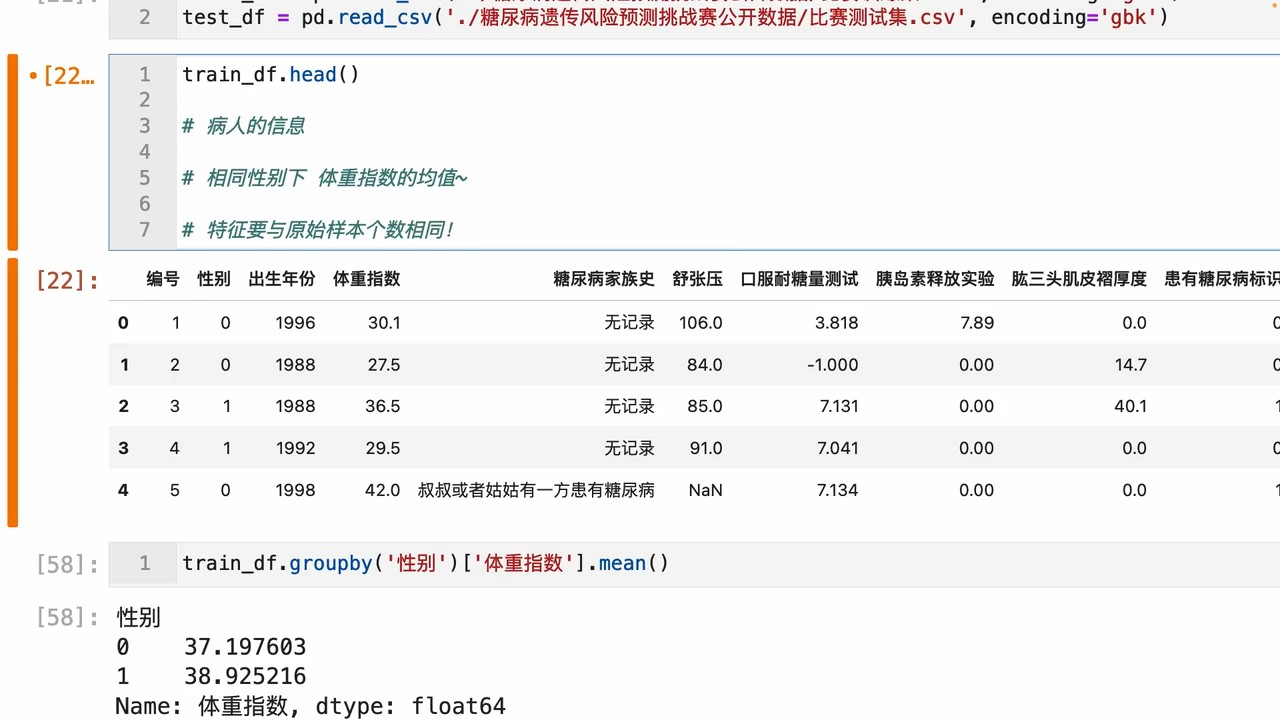
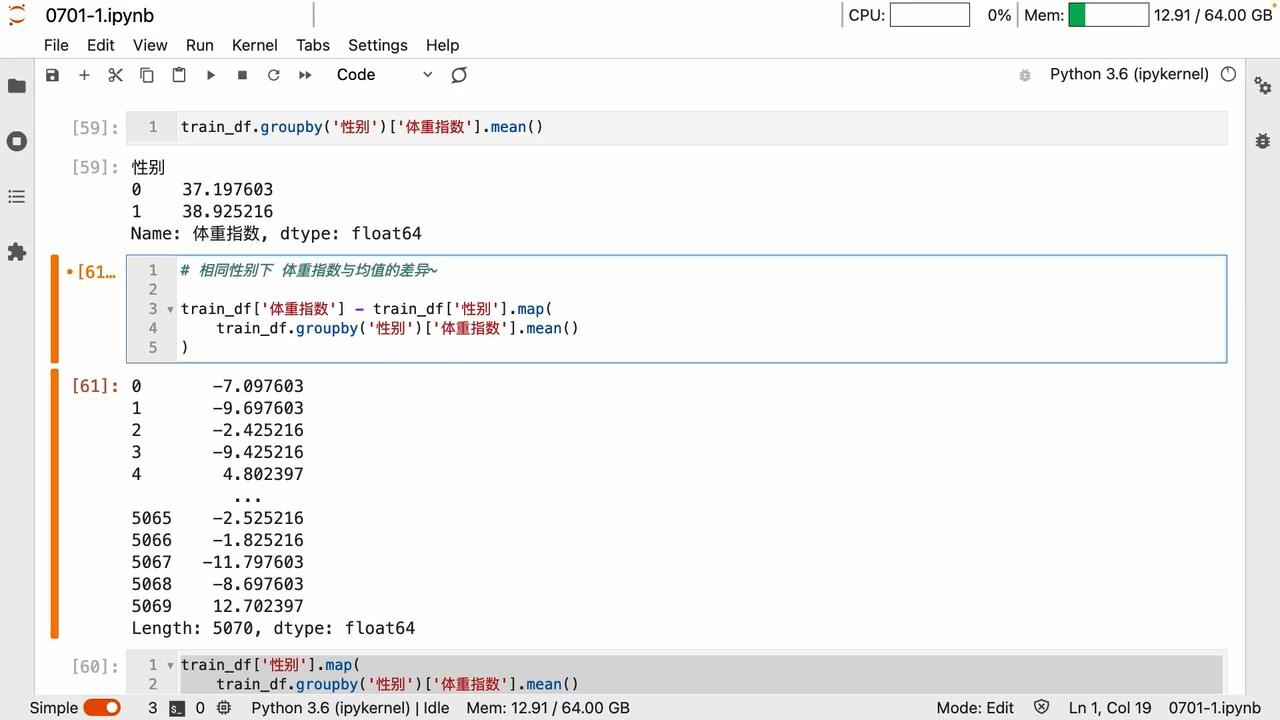
可以这样产生一列维度合适的新的特征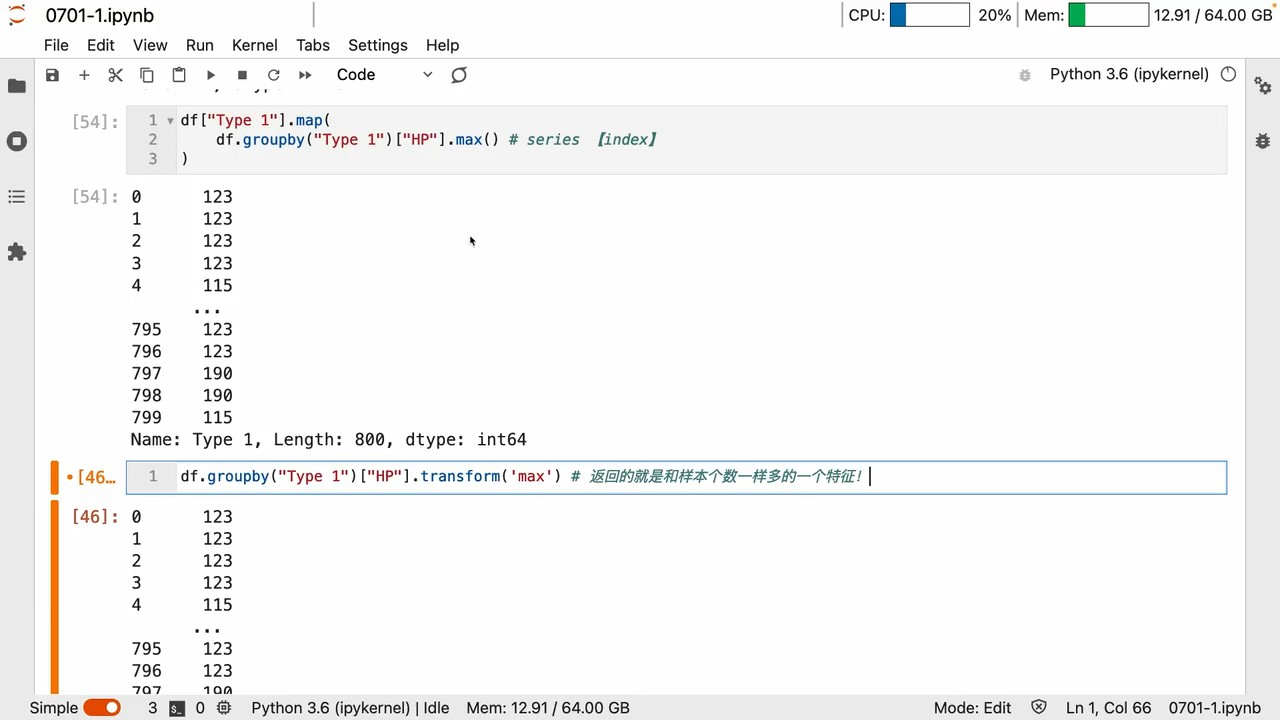
可视化
见上面步骤三的
特征工程
onehot

labelEncoder
通常的类别变量用labelEncoder即可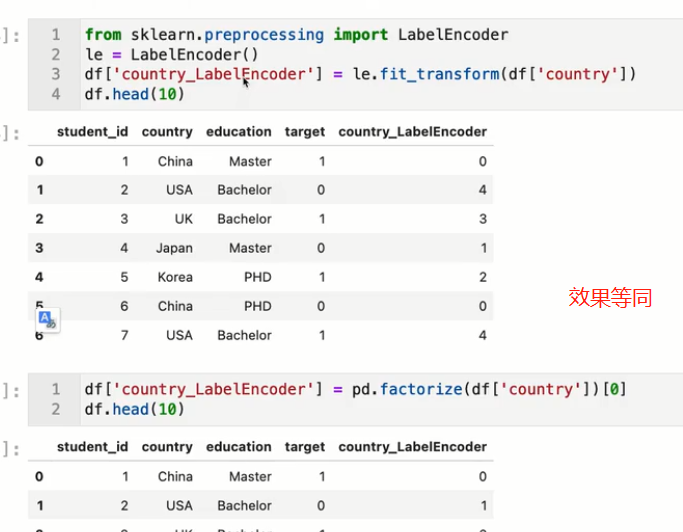
有序变量
需要保留顺序的 有序变量 用map
任务3:逻辑回归尝试
步骤1:导入sklearn中的逻辑回归;
import pandas as pd
from sklearn.linear_model import LogisticRegression
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import make_pipeline
导入对应的包
步骤1.5 数据预处理
我觉得因为家族史根据常识可以当成有序变量,而且‘叔叔或者姑姑有一方患有糖尿病’和‘叔叔或姑姑有一方患有糖尿病’其实(含义一样)是一类,如果直接用labelEncoder就会变成两类,是不对的,所以用map处理。
train_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛训练集.csv', encoding='gbk')
test_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛测试集.csv', encoding='gbk')
dict_糖尿病家族史 = {
'无记录': 0,
'叔叔或姑姑有一方患有糖尿病': 1,
'叔叔或者姑姑有一方患有糖尿病': 1,
'父母有一方患有糖尿病': 2
}
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].map(dict_糖尿病家族史)
test_df['糖尿病家族史'] = test_df['糖尿病家族史'].map(dict_糖尿病家族史)
train_df['舒张压'].fillna(89, inplace=True)
test_df['舒张压'].fillna(89, inplace=True)
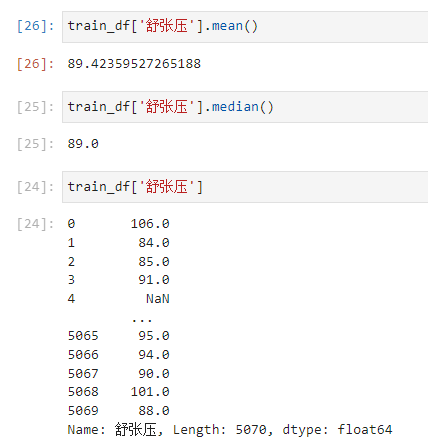
舒张压用平均值或者中位数都是89这个值这里,而且观察原来数据都是整数,所以就用89了。
补全之后,没有缺失值了。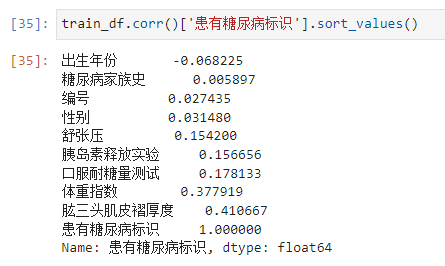
家族史和糖尿病标识之间的相关性,这么低吗。(意外,明明地球OL里面,遗传是导致糖尿病的重要因素)
步骤2:使用训练集和逻辑回归进行训练,并在测试集上进行预测;
读数据-》将有序变量转换为数值变量(利用map)
根据前面数据分析,(4.9%的缺失不至于删除),所以补全缺失值
from sklearn.linear_model import LogisticRegression as LR
from sklearn.model_selection import cross_val_score
from sklearn.feature_selection import SelectFromModel
import numpy as np
import pandas as pd
from sklearn.metrics import accuracy_score
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import make_pipeline
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score
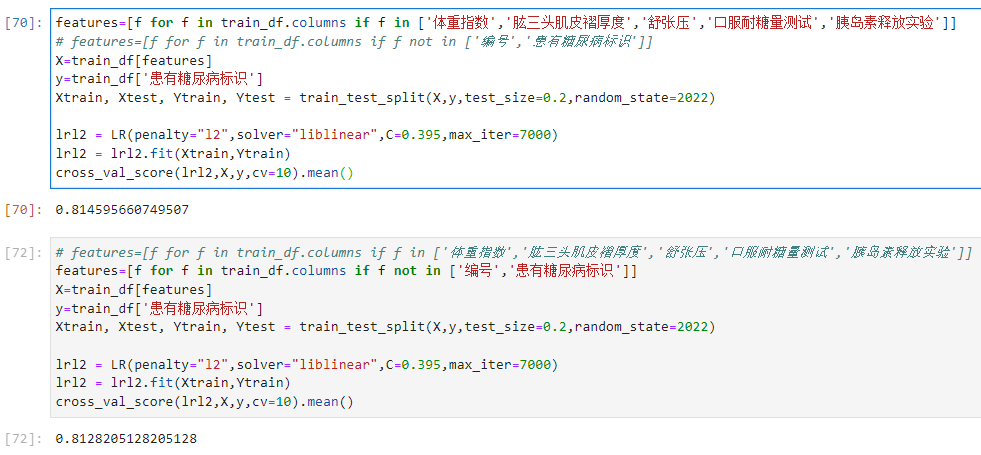
features=[f for f in train_df.columns if f not in ['编号','患有糖尿病标识','出生年份']]
X=train_df[features]
y=train_df['患有糖尿病标识']
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.2,random_state=2022)
#修正索引
for i in [Xtrain, Xtest, Ytrain, Ytest]:
i.index=range(i.shape[0])
#用图像来确认
l1 = []
l2 = []
l1test = []
l2test = []
for i in np.linspace(0.05,1,19):
lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000)
lrl1 = lrl1.fit(Xtrain,Ytrain)
l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain))
l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest))
lrl2 = lrl2.fit(Xtrain,Ytrain)
l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain))
l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest))
graph = [l1,l2,l1test,l2test]
color = ["green","black","lightgreen","gray"]
label = ["L1","L2","L1test","L2test"]
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.05,1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4)
plt.show()
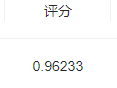
步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
我是看 l2正则化的逻辑回归,然后再测试集上的表现好一点,然后又通过缩小空间,找到了表现最好的C值,结果交上去,才71.(可能是我把 出生年份 给剔除掉的原因)也有可能识别的原因 谁知道呢
lrl2 = LR(penalty="l2",solver="liblinear",C=0.395,max_iter=1000)
lrl2 = lrl2.fit(Xtrain,Ytrain)
test_df['label'] = lrl2.predict(test_df.drop(['编号','出生年份'], axis=1))
test_df.rename({
'编号': 'uuid'}, axis=1)[['uuid', 'label']].to_csv('submit.csv', index=None)
步骤4:将训练集20%划分为验证集,在训练部分进行训练,在测试部分进行预测,调节逻辑回归的超参数;
(看了一下别人博客,也没规定l1,l2,就默认l2,然后C=1e2,然后评分就73)
步骤4:如果精度有提高,则重复步骤2和步骤3;如果没有提高,可以尝试树模型,重复步骤2、3;
#网格搜索
parameters = {
'splitter':('best','random')
,'criterion':("gini","entropy")
,"max_depth":[*range(1,10)]
# 'min_samples_leaf':[*range(1,50,5)]
# ,'min_impurity_decrease':[*np.linspace(0,0.5,20)]
}
clf = DecisionTreeClassifier(random_state=25)
GS = GridSearchCV(clf, parameters, cv=10)
GS.fit(Xtrain,Ytrain)
GS.best_params_
# GS.best_score_
#画曲线去找
tr=[]
te=[]
for i in range(40):
clf=DecisionTreeClassifier(random_state=25
,max_depth=7
,criterion="entropy"
,min_samples_leaf=i+1
)
clf=clf.fit(Xtrain,Ytrain)
score_tr=clf.score(Xtrain,Ytrain)
score_te=cross_val_score(clf,X,y,cv=10).mean()
tr.append(score_tr)
te.append(score_te)
print(max(te),te.index(max(te)))
plt.plot(range(1,41),tr,color='red',label='train')
plt.plot(range(1,41),te,color='green',label='test')
plt.xticks(range(1,41))
plt.legend()
plt.show()
提交答案
clf = DecisionTreeClassifier(random_state=25
,min_samples_leaf=25
,max_depth=7
,criterion="entropy"
)
clf.fit(Xtrain,Ytrain)
score=cross_val_score(clf,X,y,cv=10).mean()
score
任务4:特征工程(使用pandas完成)
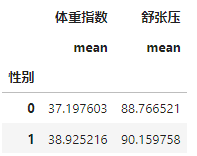
步骤1:统计每个性别对应的【体重指数】、【舒张压】平均值
train_df.groupby("性别").agg({
'体重指数':['mean'],'舒张压':['mean']})
步骤2:计算每个患者与每个性别平均值的差异;
train_df['体重指数mean']=train_df['体重指数']-train_df['性别'].map(
train_df.groupby("性别")['体重指数'].mean()
)
train_df['舒张压mean']=train_df['舒张压']-train_df['性别'].map(
train_df.groupby("性别")['舒张压'].mean()
)
从相关性上来看,差不多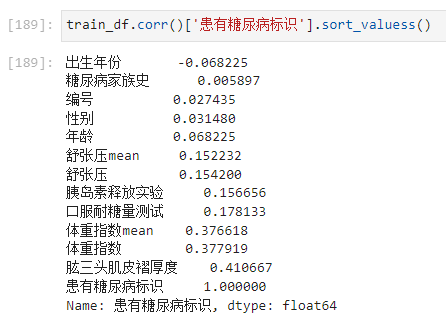
步骤3:在上述基础上将训练集20%划分为验证集,使用逻辑回归完成训练,精度是否有提高?
步骤4:思考字段含义,尝试新的特征,将你的尝试写入博客;
import pandas as pd
data={
'featureName':features,'importances':clf.feature_importances_.tolist()}
df = pd.DataFrame(data)
df=df.sort_values(by=['importances'],ascending=False)
df
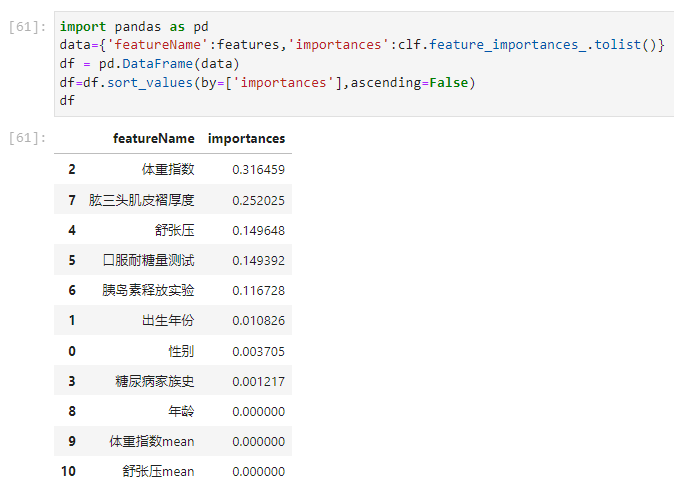
新特征 好像没啥用,决策树对他们看不上眼。
任务5:特征筛选
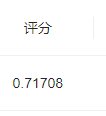
步骤1:使用树模型完成模型的训练,通过特征重要性筛选出Top5的特征;
根据上面的图片可以看出
体重指数 0.316459
肱三头肌皮褶厚度 0.252025
舒张压 0.149648
口服耐糖量测试 0.149392
胰岛素释放实验 0.116728
步骤2:使用筛选出的特征和逻辑回归进行训练,在验证集精度是否有提高?
提高了
步骤3:如果有提高,为什么?如果没有提高,为什么?

划重点了,考的好了一点点。
但这肯定不是明显提高,模型到极限了,逻辑回归也就这了。
步骤4:将你的尝试写入博客;
任务6:高阶树模型
import pandas as pd
import numpy as np
import seaborn as sns
from sklearn.model_selection import KFold
import lightgbm as lgb
# 读取数据
train_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛训练集.csv', encoding='gbk')
test_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛测试集.csv', encoding='gbk')
# 基础特征工程
train_df['体重指数_round'] = train_df['体重指数'] // 10
test_df['体重指数_round'] = train_df['体重指数'] // 10
# //地板除 Python中两个斜杠即双斜杠(//)表示地板除,即先做除法(/),然后向下取整(floor)。
train_df['口服耐糖量测试'] = train_df['口服耐糖量测试'].replace(-1, np.nan)
test_df['口服耐糖量测试'] = test_df['口服耐糖量测试'].replace(-1, np.nan)
# 这个特征里面有-1 懵逼了
dict_糖尿病家族史 = {
'无记录': 0,
'叔叔或姑姑有一方患有糖尿病': 1,
'叔叔或者姑姑有一方患有糖尿病': 1,
'父母有一方患有糖尿病': 2
}
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].map(dict_糖尿病家族史)
test_df['糖尿病家族史'] = test_df['糖尿病家族史'].map(dict_糖尿病家族史)
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].astype('category')
test_df['糖尿病家族史'] = train_df['糖尿病家族史'].astype('category')
train_df['性别'] = train_df['性别'].astype('category')
test_df['性别'] = train_df['性别'].astype('category')
# 这个转换为类别是有什么特别的含义吗
train_df['年龄'] = 2022 - train_df['出生年份']
test_df['年龄'] = 2022 - test_df['出生年份']
#前面操作过了,可是没什么用
train_df['口服耐糖量测试_diff'] = train_df['口服耐糖量测试'] - train_df.groupby('糖尿病家族史').transform('mean')['口服耐糖量测试']
test_df['口服耐糖量测试_diff'] = test_df['口服耐糖量测试'] - test_df.groupby('糖尿病家族史').transform('mean')['口服耐糖量测试']
#原来这个特征要操作,而不是体重和舒张压
步骤1:安装LightGBM,并学习基础的使用方法;
步骤2:将训练集20%划分为验证集,使用LightGBM完成训练,精度是否有提高?
用coogle老师给的模板代码跑一下
# 模型交叉验证
def run_model_cv(model, kf, X_tr, y, X_te, cate_col=None):
train_pred = np.zeros( (len(X_tr), len(np.unique(y))) )
test_pred = np.zeros( (len(X_te), len(np.unique(y))) )
cv_clf = []
for tr_idx, val_idx in kf.split(X_tr, y):
x_tr = X_tr.iloc[tr_idx]; y_tr = y.iloc[tr_idx]
x_val = X_tr.iloc[val_idx]; y_val = y.iloc[val_idx]
call_back = [
lgb.early_stopping(50),
]
eval_set = [(x_val, y_val)]
model.fit(x_tr, y_tr, eval_set=eval_set, callbacks=call_back, verbose=-1)
cv_clf.append(model)
train_pred[val_idx] = model.predict_proba(x_val)
test_pred += model.predict_proba(X_te)
test_pred /= kf.n_splits
return train_pred, test_pred, cv_clf
```python
clf = lgb.LGBMClassifier(
max_depth=3,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=0.1,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
train_df.drop(['编号', '患有糖尿病标识'], axis=1),
train_df['患有糖尿病标识'],
test_df.drop(['编号'], axis=1),
)
print((train_pred.argmax(1) == train_df['患有糖尿病标识']).mean())
test_df['label'] = test_pred.argmax(1)
test_df.rename({
'编号': 'uuid'}, axis=1)[['uuid', 'label']].to_csv('submit.csv', index=None)
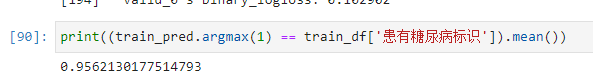
步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
步骤4:尝试调节搜索LightGBM的参数;
看了不同深度的参数的影响
lgbtrain = []
lgbtest = []
for i in range(10):
clf = lgb.LGBMClassifier(
max_depth=i+1,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=0.1,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
Xtrain,
Ytrain,
Xtest,
)
lgbtrain.append((train_pred.argmax(1) == Ytrain).mean())
lgbtest.append((test_pred.argmax(1) == Ytest).mean())
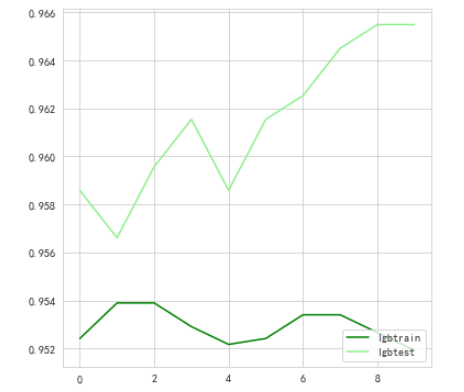
看了不同学习率的参数的影响
lgbtrain = []
lgbtest = []
# for i in np.linspace(0.39,0.40,19):
for i in np.linspace(0.001,0.1,19):
clf = lgb.LGBMClassifier(
max_depth=2,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=i,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
Xtrain,
Ytrain,
Xtest,
)
lgbtrain.append((train_pred.argmax(1) == Ytrain).mean())
lgbtest.append((test_pred.argmax(1) == Ytest).mean())
graph = [lgbtrain,lgbtest]
color = ["green","lightgreen"]
label = ["lgbtrain","lgbtest"]
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.001,0.1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4)
plt.show()
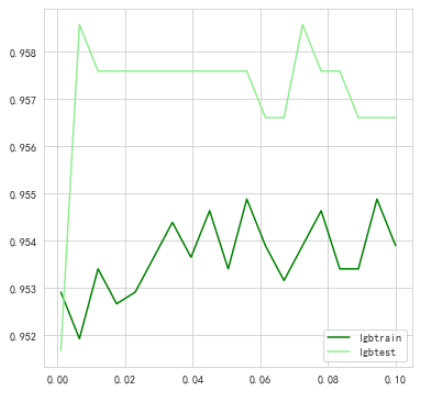
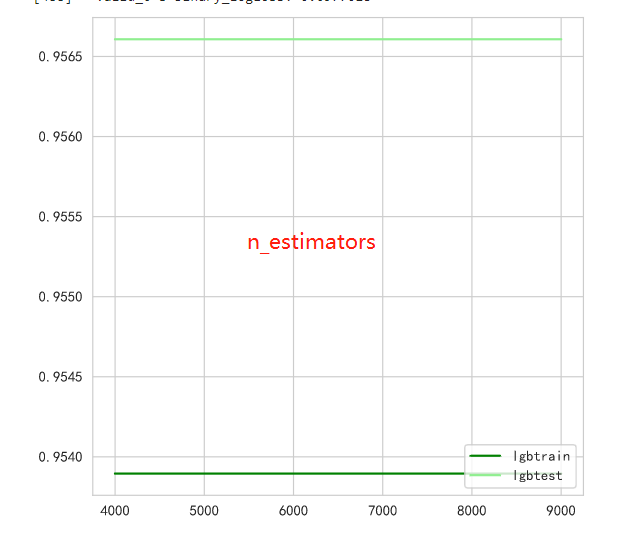
步骤5:将步骤4调参之后的模型从新训练,将最新预测的结果文件提交到比赛,截图分数;
蚌埠住了,更低了
任务7:多折训练与集成
可以参考 大佬的https://blog.csdn.net/qq_39473431/article/details/125478123
还有大佬提及的 https://www.kaggle.com/code/arthurtok/introduction-to-ensembling-stacking-in-python/notebook
步骤1:使用KFold完成数据划分;
KFold划分数据集的原理:根据n_split直接进行划分。已经划分过了
步骤2:使用StratifiedKFold完成数据划分;
StratifiedKFold划分数据集的原理:划分后的训练集和验证集中类别分布尽量和原数据集一样
使用
数据竞赛中的实践
理论
调参
步骤3:使用StratifiedKFold配合LightGBM完成模型的训练和预测
coggle给的范例是KFold的,修改相应代码即可
步骤4:在步骤3训练得到了多少个模型,对测试集多次预测,将最新预测的结果文件提交到比赛,截图分数;
步骤4:使用交叉验证训练5个机器学习模型(svm、lr等),使用stacking完成集成,将最新预测的结果文件提交到比赛,截图分数;
集成
做一个好的模型,首先要有好的数据,数据的质量决定了你模型效果的上限;其次,要做好特征工程,在无法改变数据质量的条件下,特征工程是重中之重;最后,建立模型。
有三种主流的模型思想,一种是Bagging,代表模型是随机森林;一种是Boosting,代表模型是GBDT、Xgboost、lightGBM;还一种是stacking或blending。前2种模型可以调参。
总结
可以像这样
把难点和过程都叙述出来
边栏推荐
- [WeChat applet] WXML template syntax - conditional rendering
- upload upload pictures to Tencent cloud, how to upload pictures
- human weakness
- mutillidae download and installation
- server disk array
- 【无标题】
- 重新审视分布式系统:永远不会有完美的一致性方案……
- Returned object not currently part of this pool
- Mysql5.7 二进制 部署
- Develop a highly fault-tolerant distributed system
猜你喜欢
Wise Force Deleter强制删除工具

【cesium】加载并定位 3D Tileset

The difference between span tag and p
WPF中DataContext作用

Structured light 3D reconstruction (1) Striped structured light 3D reconstruction
upload upload pictures to Tencent cloud, how to upload pictures
Develop a highly fault-tolerant distributed system
The underlying mechanism of the class
Dephi reverse tool Dede exports function name MAP and imports it into IDA
『递归』递归概念与典型实例
随机推荐
Dephi reverse tool Dede exports function name MAP and imports it into IDA
1068 Find More Coins
『递归』递归概念与典型实例
"PHP8 Beginner's Guide" A brief introduction to PHP
雷克萨斯lm的安全性到底体现在哪里?一起来看看吧
Community Sharing|Tencent Overseas Games builds game security operation capabilities based on JumpServer
jvm 三 之堆与栈
MySQL基础(一)---基础认知及操作
Algorithms - ones and zeros (Kotlin)
2023年信息与通信工程国际会议(JCICE 2023)
redis事务
使用二维码解决固定资产管理的难题
Distributed systems revisited: there will never be a perfect consistency scheme...
重新审视分布式系统:永远不会有完美的一致性方案……
Detailed explanation of each module of ansible
OFDM 十六讲 5 -Discrete Convolution, ISI and ICI on DMT/OFDM Systems
类的底层机制
u-boot debugging and positioning means
CAP+BASE
【cesium】加载并定位 3D Tileset