当前位置:网站首页>(五)R语言入门生物信息学——ORF和序列分析
(五)R语言入门生物信息学——ORF和序列分析
2022-07-06 09:17:00 【EricFrenzy】
注:本博客旨在分享个人学习心得,有不规范之处请多多包涵!
概念介绍
在人体内,为了表达DNA上的基因,这个基因包含的DNA在被转录为pre-mRNA后经过进一步处理成为成熟的mRNA,mRNA紧接着会被核糖体用来合成蛋白质,从而控制生物体的反应。在mRNA上,每三个碱基组成一个密码子,对应一种氨基酸。下图为密码子与氨基酸的对照表: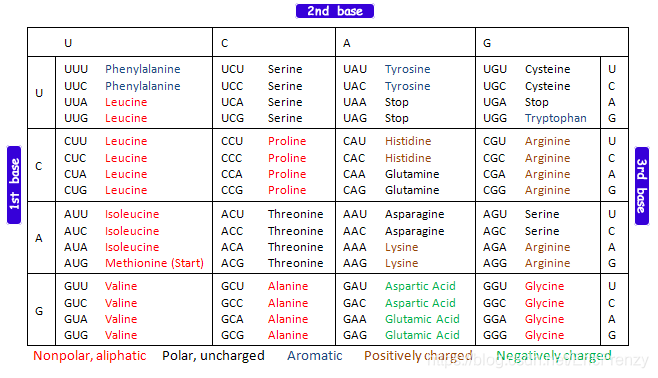
要合成一个正常的蛋白质,mRNA序列上的两端需要分别有一个起始密码子(图上标为start)和一个终止密码子(图上标为stop)。但在DNA上有许多这样子的起始和终止密码子,产生很多种不同的序列组合。为了在DNA上找到所有可能的能用来制作某种蛋白质的序列组合,我们用开放阅读框(ORF,Open Reading Frame)来找到所有具有编码蛋白质潜能的序列。
找ORF的代码实现
在R语言中找ORF的程序流程如下: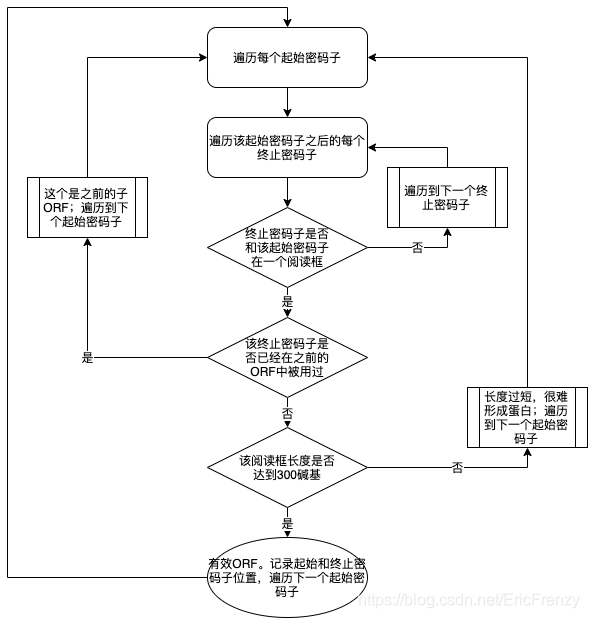
下面是具体代码:
findORF <- function(seq){
#传入参数为DNA序列,注意方向一定要是5'到3'
findStartCodons <- function(seq){
#找起始密码子的函数
startcodons <- numeric(0) #创建空函数
k <- 1
for(i in 1:(length(seq)-5)){
#以密码子的第一位碱基位置计算,最后五位无需检查,因为长度过短
if(seq[i] == "a" && seq[i+1] == "t" && seq[i+2] == "g"){
#ATG对应起始密码子
startcodons[k] <- i #记录位置
k <- k + 1 #位置下标加一
}
}
return(startcodons) #返回结果
}
findStopCodons <- function(seq){
#找终止密码子的函数
stopcodons <- numeric(0) #创建空函数
k <- 1
for(i in 1:(length(seq)-2)){
#以密码子的第一位碱基位置计算
if((seq[i] == "t" && seq[i+1] == "a" && seq[i+2] == "a") || (seq[i] == "t" && seq[i+1] == "a" && seq[i+2] == "g") || (seq[i] == "t" && seq[i+1] == "g" && seq[i+2] == "a")){
#TAA TAG TGA对应终止密码子
stopcodons[k] <- i #记录位置
k <- k + 1 #位置下标加一
}
}
return(stopcodons) #返回结果
}
startcodon <- findStartCodons(seq) #找到所有的起始密码子
stopcodon <- findStopCodons(seq) #找到所有的终止密码子
usedStop <- numeric(0) #记录用过的终止密码子
ORFs <- character(0) #记录有效开放阅读框
k <- 1
for(i in startcodon){
#遍历所有起始密码子
for(j in stopcodon){
#遍历所有终止密码子
if((j-i)%%3==0 && j > i){
#如果在一个阅读框内,即两个密码子之间的位置为3的整数
if(j %in% usedStop){
#如果终止密码子被用过
break #跳出这次循环,到下一个起始密码子
}else if(j-i < 300){
#如果密码子之间的序列长度过短
break #同上
}else{
ORFs[k] <- paste(i, "to", j) #生成字符串,记录的结果如"1 to 3001"
usedStop[k] <- j #记录用过的终止密码子
k <- k + 1 #位置下标加一
break #跳出本次循环,到下一个起始密码子
}
}
}
}
return(ORFs) #返回结果
}
这种找ORF的算法比较简单快速,但相应地准确度会有所下降。在NCBI官网有更准确的算法。
结束语
在找到ORF后,就能将该ORF与数据库中的已知序列进行比对,从而预测该物种基因的组成与功能等有用信息。下次将会介绍Needleman-Wunsch这一序列全局比对算法,敬请期待!和有任何问题或想法欢迎留言和评论!
边栏推荐
猜你喜欢
arduino UNO R3的寄存器写法(1)-----引脚电平状态变化

Machine learning -- decision tree (sklearn)
OPPO VOOC快充电路和协议

History object

Basic operations of databases and tables ----- creating data tables
Characteristics, task status and startup of UCOS III
JS 函数提升和var变量的声明提升
RT-Thread的main线程“卡死”的一种可能原因及解决方案
电商数据分析--薪资预测(线性回归)
open-mmlab labelImg mmdetection
随机推荐
Reno7 60W超级闪充充电架构
RuntimeError: cuDNN error: CUDNN_ STATUS_ NOT_ INITIALIZED
【ESP32学习-1】Arduino ESP32开发环境搭建
基於Redis的分布式ID生成器
map文件粗略分析
Bubble sort [C language]
open-mmlab labelImg mmdetection
VIM command line notes
JS變量類型以及常用類型轉換
Redis 缓存更新策略,缓存穿透、雪崩、击穿问题
inline详细讲解【C语言】
GCC compilation options
C语言函数之可变参数原理:va_start、va_arg及va_end
Pytoch temperature prediction
List and set
Symbolic representation of functions in deep learning papers
Knowledge summary of request
MP3mini播放模块arduino<DFRobotDFPlayerMini.h>函数详解
Stm32f1+bc20+mqtt+freertos system is connected to Alibaba cloud to transmit temperature and humidity and control LED lights
History object